
ADN
?? propos de ce ??coles s??lection Wikipedia
SOS Enfants, un organisme de bienfaisance de l'??ducation , a organis?? cette s??lection. SOS Children travaille dans 45 pays africains; pouvez-vous aider un enfant en Afrique ?

L'acide d??soxyribonucl??ique (ADN) est une mol??cule qui code les g??n??tiques instructions utilis??es dans le d??veloppement et le fonctionnement de tous les vivants connus organismes et de nombreux virus . Avec L'ARN et des prot??ines , de l'ADN est l'un des trois principaux macromol??cules essentiel pour toutes les formes connues de la vie . L'information g??n??tique est cod??e par une s??quence de nucleotides ( guanine, ad??nine, thymine, et cytosine) enregistr??e en utilisant les lettres G, A, T, C et plus mol??cules d'ADN double brin sont h??lices, compos?? de deux longues polym??res d'unit??s simples appel??s des nucl??otides, des mol??cules avec squelettes en alternance sucres ( d??soxyribose) et (groupes phosphate li??s ?? l'acide phosphorique), avec le nucl??obases (G, A, T, C) associ??es aux sucres. L'ADN est bien adapt?? pour le stockage de l'information biologique, ??tant donn?? que le squelette d'ADN r??sistant ?? la coupure et la structure double brin permet la mol??cule avec un haut-copie de l'information cod??e.
Ces deux volets dirig??s dans des directions oppos??es les unes aux autres et sont donc anti-parall??le, une ??pine dorsale ??tant de 3 '(de trois prime) et l'autre 5' (cinq premier). Il se agit de la direction le carbone 3??me et 5??me sur la mol??cule de sucre est confront??e. Attach??s ?? chaque sucre est l'un des quatre types de mol??cules appel??es nucl??obases (officieusement, bases). C'est le s??quence de ces quatre nucl??obases le long du squelette qui code l'information. Ces informations sont lues ?? l'aide du code g??n??tique , qui sp??cifie la s??quence des acides amin??s au sein de prot??ines. Le code est lu en copiant segments d'ADN dans le apparent?? ARN d'acide nucl??ique dans un processus appel?? transcription.
Dans les cellules, l'ADN est organis?? en structures appel??es longues chromosomes. Pendant la division cellulaire ces chromosomes sont dupliqu??s dans le processus de r??plication de l'ADN, chaque cellule fournissant son propre ensemble complet de chromosomes. Les organismes eucaryotes ( animaux de , plantes , champignons , et protistes) magasin le plus de leur ADN ?? l'int??rieur du noyau cellulaire et une partie de leur ADN en organites tels que les mitochondries ou chloroplastes. En revanche, procaryotes ( bact??ries et arch??es) stocker leur ADN seulement dans le cytoplasme. Dans les chromosomes, des prot??ines telles que la chromatine histones compacte et organiser ADN. Ces structures compactes guident les interactions entre l'ADN et d'autres prot??ines, en aidant contr??le quelles parties de l'ADN sont transcrites.
Propri??t??s

L'ADN est une longue polym??re compos?? de motifs r??p??titifs appel??s nucl??otides. ADN a ??t?? identifi?? et isol?? par Friedrich Miescher et la structure en double h??lice de l'ADN a ??t?? d??couvert par James D. Watson et Francis Crick . La structure de l'ADN de toutes les esp??ces comprend deux cha??nes h??lico??dales chacun enroul?? autour d'un m??me axe, et chacune avec un pas de 34 (3,4 Angstr??ms nanom??tres) et un rayon de 10 Angstr??ms (1,0 nanom??tres). Selon une autre ??tude, lorsqu'elle est mesur??e dans une solution particuli??re, la cha??ne d'ADN mesur??e 22 ?? 26 angstr??ms large (02/02 au 02/06 nanom??tres), et une unit?? de nucleotides ??valu??s 3,3 ?? (0,33 nm) de longueur. Bien que chaque unit?? r??p??titive individuelle est tr??s faible, les polym??res d'ADN peuvent ??tre tr??s grandes mol??cules contenant des millions de nucleotides. Par exemple, la plus grande humain chromosome, le chromosome num??ro 1, est d'environ 220 millions paires de bases de long.
Dans les organismes vivants ne existe g??n??ralement pas de l'ADN en tant que mol??cule unique, mais plut??t comme une paire de mol??cules qui sont maintenus ??troitement ensemble. Ces deux longs brins enlacent comme des vignes, dans la forme d'un double h??lice. Les r??p??titions de nucl??otides contiennent ?? la fois le segment de la cha??ne principale de la mol??cule, qui maintient ensemble la cha??ne, et une nucl??obase qui coop??re avec l'autre brin d'ADN dans l'h??lice. Une base nucl??ique li??e ?? un sucre est appel??e un et une base de nucl??oside li?? ?? un sucre et un ou plusieurs groupes phosphate est appel??e nucl??otide. Polym??re comprenant plusieurs nucleotides li??s (comme dans l'ADN) est appel??e une polynucl??otide.
L'??pine dorsale du brin d'ADN est fait de l'alternance de phosphate et de sucre r??sidus. Le sucre est dans l'ADN 2-d??soxyribose, qui est un pentoses (cinq carbone sucre). Les sucres sont reli??es entre elles par des groupes phosphate qui forment des liaisons phosphodiester entre les troisi??me et cinqui??me carbone des atomes de cycles de sucres adjacents. Ces asym??trique obligations signifierait un brin d'ADN a une direction. Dans une double h??lice la direction de nucl??otides dans un brin est oppos??e ?? leur direction dans l'autre brin: les brins sont antiparall??les. Les extr??mit??s asym??triques de brins d'ADN sont appel??es 5 '(cinq Premier) et 3 '(trois prime) se termine, ?? l'extr??mit?? 5' ayant un groupe phosphate terminal et l'extr??mit?? 3 'd'un groupe hydroxyle terminal. Une diff??rence majeure entre l'ADN et l'ARN est le sucre, avec le 2-d??soxyribose dans l'ADN ??tant remplac?? par le sucre pentose alternatif ribose de l'ARN.

La double h??lice de l'ADN est stabilis??e principalement par deux forces: des liaisons hydrog??ne entre les nucl??otides et interactions empilement de bases entre nucl??obases aromatiques. Dans l'environnement aqueux de la cellule, le conjugu?? π liaisons de bases de nucl??otides align??s perpendiculairement ?? l'axe de la mol??cule d'ADN, ce qui r??duit leur interaction avec le couche de solvatation et donc, l' ??nergie libre de Gibbs . Les quatre bases de l'ADN sont ad??nine (en abr??g?? A), cytosine (C), guanine (G) et thymine (T). Ces quatre bases sont fix??s au sucre / phosphate pour former le nucl??otidique compl??te, comme le montre par ad??nosine monophosphate.
Classement nucl??obases
Les nucl??obases sont class??s en deux types: le purines, A et G, ??tant fusionn??es ?? cinq et ?? six cha??nons les compos??s h??t??rocycliques, et le les pyrimidines, les cycles ?? six cha??nons C et T. Un cinqui??me nucl??obase pyrimidine, uracile (U), a g??n??ralement lieu de thymine dans l'ARN et diff??re de la thymine par un manque groupe m??thyle sur son anneau. En plus de l'ARN et de l'ADN d'un grand nombre de artificielle des analogues d'acides nucl??iques ont ??galement ??t?? cr????s pour ??tudier les propri??t??s des acides nucl??iques, ou pour une utilisation en biotechnologie.
L'uracile est pas habituellement trouv?? dans l'ADN, qui a lieu uniquement en tant que produit de d??gradation de la cytosine. Cependant, dans un certain nombre de bact??riophages - bacteriophages de Bacillus subtilis PBS1 et PBS2 et Yersinia piR1-37 bacteriophage - thymine est remplac??e par l'uracile. Une forme modifi??e (b??ta-d-glucopyranosyloxymethyluracil) se trouve ??galement dans un certain nombre d'organismes: les flagell??s Diplot??ne et Euglena, et tout le Biosynth??se des genres kin??toplastide J se produit en deux ??tapes: dans la premi??re ??tape une thymidine dans l'ADN sp??cifique est convertie en hydroxymethyldeoxyuridine; dans la seconde HOMedU est glycosyl??e pour former des prot??ines qui se lient sp??cifiquement J. de cette base ont ??t?? identifi??s. Ces prot??ines semblent ??tre des parents ??loign??s de l'oncog??ne TET1 qui est impliqu??e dans la pathogen??se de la leuc??mie my??lo??de aigu??. J semble agir comme un signal de terminaison pour L'ARN polym??rase II.

Grooves
Brins h??lico??daux jumeaux forment l'??pine dorsale de l'ADN. Un autre double h??lice peut ??tre trouv?? tra??ant les espaces, ou des rainures, entre les brins. Ces vides sont adjacentes aux paires de bases et peuvent fournir une site de liaison. Comme les fils ne sont pas situ??s sym??triquement par rapport ?? l'autre, les rainures sont de taille in??gale. Une rainure, le grand sillon, est 22 une large et l'autre, le petit sillon, est 12 une large. L'??troitesse du sillon veut dire que les bords des bases sont plus accessibles dans le grand sillon. En cons??quence, les prot??ines comme les facteurs de transcription qui peuvent se lier ?? des s??quences sp??cifiques dans l'ADN double brin font g??n??ralement contacts aux c??t??s des bases expos??es dans le grand sillon. Cette situation varie en conformations inhabituelles de l'ADN dans la cellule (voir ci-dessous), mais les majeures et mineures rainures sont toujours nomm??s pour refl??ter les diff??rences dans la taille que l'on pouvait voir si l'ADN est tordu de nouveau dans la forme ordinaire B.
Base de jumelage
Dans une double h??lice d'ADN, chaque type de nucl??obases sur les obligations d'un brin avec un seul type de nucl??obases sur l'autre brin. Cela se appelle compl??mentaires appariement base. Ici, forment purines des liaisons hydrog??ne avec des pyrimidines, ?? l'ad??nine ?? thymine liaison seulement dans deux liaisons hydrog??ne, et la liaison ?? la cytosine guanine dans seulement trois liaisons hydrog??ne. Cet agencement de deux nucleotides liant ensemble ?? travers la double h??lice est appel??e une paire de bases. Comme liaisons hydrog??ne ne sont pas covalente, ils peuvent ??tre bris?? et rejoint relativement facilement. Les deux brins d'ADN en double h??lice peuvent donc ??tre s??par??s comme une fermeture ?? glissi??re, soit par une force m??canique ou haute temp??rature . A la suite de cette compl??mentarit??, toutes les informations de la s??quence ?? double brin d'une h??lice d'ADN est dupliqu?? sur chaque brin, qui est essentiel dans la r??plication de l'ADN. En effet, cette interaction r??versible et sp??cifique entre des paires de bases compl??mentaires est essentiel pour toutes les fonctions de l'ADN dans les organismes vivants.
 |
 |
Les deux types de paires de base forment des nombres diff??rents de liaisons hydrog??ne, AT formant deux liaisons hydrog??ne, et GC former trois liaisons hydrog??ne (voir les figures, ?? droite). ADN avec haute GC-contenu est plus stable que l'ADN ?? faible contenu en GC.
Comme indiqu?? plus haut, la plupart des mol??cules d'ADN sont en fait deux brins polym??riques li??s entre eux, de fa??on h??lico??dale par des liaisons non covalentes; cette structure double brin (ADNdb) est maintenu en grande partie par la base intrabrin interactions d'empilement, qui sont les plus forts G, C empile. Les deux brins peuvent se s??parer - un processus connu sous le nom de fusion - pour former deux mol??cules ADN simple brin. Fusion se produit lorsque les conditions favorisent ssADN; ces conditions sont une temp??rature ??lev??e, faible teneur en sel et pH ??lev?? (pH bas fond ??galement de l'ADN, mais puisque l'ADN est instable en raison de d??purination acide, pH bas est rarement utilis??).
La stabilit?? de la forme ADN double brin d??pend non seulement de la GC-teneur (% G, C paires de bases), mais aussi sur la s??quence (s??quence depuis empilement est sp??cifique) ainsi que la longueur (mol??cules plus longues sont plus stables). La stabilit?? peut ??tre mesur??e de diverses mani??res; une voie commune est la "temp??rature de fusion", qui est la temp??rature ?? laquelle 50% des mol??cules ds sont converties en mol??cules de ss; la temp??rature de fusion d??pend de la force ionique et la concentration de l'ADN. En cons??quence, il est ?? la fois le pourcentage de paires de bases GC et la longueur totale d'une double h??lice d'ADN qui d??termine la force de l'association entre les deux brins d'ADN. H??lices d'ADN longues avec un GC-haute teneur ont brins solides interagissant, tandis courtes h??lices avec teneur ??lev??e en AT ont brins faibles interagissant. En biologie, des parties de la double h??lice d'ADN qui doivent se s??parer facilement, par exemple le TATAAT Pribnow bo??te dans certains promoteurs, ont tendance ?? avoir une teneur ??lev??e en AT, ce qui rend les brins plus faciles ?? s??parer.
Dans le laboratoire, la force de cette interaction peut ??tre mesur??e en trouvant la temp??rature n??cessaire pour briser les liaisons hydrog??ne, leur temp??rature de fusion (??galement appel??e valeur T m). Lorsque toutes les paires de bases dans un ADN ?? double h??lice masse fondue, les brins se s??parent et existent en solution sous forme de deux mol??cules enti??rement ind??pendants. Ces mol??cules d'ADN simple brin (ADNsb) ne ont pas de forme commune unique, mais certaines conformations sont plus stables que d'autres.
Sens et antisens
Une s??quence d'ADN est appel?? "sens" si sa s??quence est la m??me que celle d'un messager copie d'ARN qui est traduit en prot??ine. La s??quence sur le brin oppos?? est appel?? la s??quence "anti-sens". Les deux s??quences sens et antisens peuvent exister sur diff??rentes parties d'un m??me brin d'ADN (ce est ?? dire les deux brins contiennent ?? la fois des s??quences sens et antisens). Dans les deux procaryotes et les eucaryotes, des s??quences d'ARN antisens sont produits, mais les fonctions de ces ARN ne sont pas tout ?? fait clair. Une proposition est que les ARN antisens sont impliqu??s dans la r??gulation l'expression g??nique par ARN-ARN appariement de bases.
Quelques-unes des s??quences d'ADN chez les procaryotes et les eucaryotes, et en plus des plasmides et des virus , brouiller la distinction entre les brins sens et antisens ayant par g??nes se chevauchant. Dans ces cas, certaines s??quences d'ADN ne double devoir, codant pour une prot??ine lors de la lecture un brin long, et une seconde prot??ine lorsqu'elle est lue dans la direction oppos??e sur l'autre brin. Dans les bact??ries , ce chevauchement peut ??tre impliqu??e dans la r??gulation de la transcription du g??ne, alors que dans les virus, les g??nes se chevauchant augmenter la quantit?? d'informations qui peut ??tre cod??e dans le petit g??nome viral.
Superenroulement
L'ADN peut ??tre tordu comme une corde dans un processus appel?? Surenroulement de l'ADN. Avec l'ADN dans son ??tat "d??tendue", un brin entoure g??n??ralement l'axe de la double h??lice une fois tous les 10,4 paires de bases, mais si l'ADN est tordu les brins devenir plus serr?? ou plus l??che des plaies. Si l'ADN est torsad?? dans la direction de l'h??lice, ce est surenroulement positif, et les bases sont maintenus plus ??troitement ensemble. Se ils sont tordus dans la direction oppos??e, ce est surenroulement n??gatif, et les bases se s??parer plus facilement. Dans la nature, la plupart de l'ADN a l??g??re surenroulement n??gatif qui est introduit par enzymes appel??s topoisom??rases. Ces enzymes sont ??galement n??cessaires pour soulager les contraintes de torsion introduits dans les brins d'ADN au cours de processus tels que transcription et r??plication de l'ADN.

Structures d'ADN Autres
ADN existe dans de nombreux possible qui comprennent des conformations A-ADN, ADN-B, et Formes Z-ADN, bien que, seul B-ADN et ADN-Z ont ??t?? directement observ??es dans les organismes fonctionnels. La conformation de l'ADN qui adopte d??pend du niveau d'hydratation, la s??quence d'ADN, la quantit?? et la direction de surenroulement, les modifications chimiques des bases, le type et la concentration de m??tal ions , ainsi que la pr??sence de polyamines en solution.
Les premiers rapports publi??s de A-DNA Diffraction des rayons X patterns- et aussi B-DNA - utilis?? analyses bas??es sur Patterson transforme pr??vu que seulement une quantit?? limit??e d'informations de structure de fibres orient??es d'ADN. Une analyse alternative a ensuite ??t?? propos?? par Wilkins et al., En 1953, pour le B-vivo de l'ADN diffraction dans X-ray / diffusion des mod??les de fibres d'ADN fortement hydrat??es en termes de places de fonctions de Bessel . Dans le m??me journal, James D. Watson et Francis Crick ont pr??sent?? leur analyse de mod??lisation mol??culaire des motifs de diffraction des rayons X ?? l'ADN sugg??rent que la structure est une double h??lice.
Bien que le formulaire `B-DNA?? est plus courante dans les conditions r??gnant dans les cellules, ce ne est pas une conformation bien d??finie, mais une famille de conformations d'ADN, qui se posent aux niveaux d'hydratation ??lev??s pr??sents dans les cellules vivantes. Les motifs de diffraction des rayons X correspondant et de diffusion sont caract??ristiques des mol??culaire paracrystals avec un degr?? significatif de troubles.
Par rapport ?? l'ADN-B, la forme A-ADN est une spirale ?? droite plus large, avec une large rainure mineure peu profonde et, une gorge profonde majeure ??troite. La forme A se produit dans des conditions non physiologiques dans des ??chantillons partiellement d??shydrat??s de l'ADN, tandis que dans la cellule, il peut ??tre produit dans les appariements hybrides d'ADN et d'ARN brins, ainsi que dans les complexes enzyme-ADN. Les segments d'ADN, o?? les bases ont ??t?? modifi??s chimiquement par m??thylation peut subir un plus grand changement de conformation et d'adopter le Forme Z. Ici, les brins tourner autour de l'axe d'h??lice dans un gaucher spirale, ?? l'oppos?? de la forme B plus fr??quent. Ces structures inhabituelles peuvent ??tre reconnues par les prot??ines de liaison Z-ADN sp??cifiques et peuvent ??tre impliqu??s dans la r??gulation de la transcription.
La chimie de l'ADN suppl??ant
Pour un certain nombre d'ann??es exobiologistes ont propos?? l'existence d'un biosph??re de l'ombre, une biosph??re microbienne postul?? de la Terre qui utilise radicalement diff??rents processus biochimiques et mol??culaires que la vie actuellement connu. Une des propositions ??tait l'existence de formes de vie qui utilisent arsenic au lieu de phosphore dans l'ADN. Un rapport en 2010 de la possibilit?? de la bact??rie GFAJ-1, a ??t?? annonc??, mais la recherche a ??t?? contest??, et les donn??es sugg??rent la bact??rie emp??che activement l'incorporation de l'arsenic dans le squelette de l'ADN et d'autres biomol??cules.
Structures quadruplex
Aux extr??mit??s des chromosomes lin??aires sont des r??gions sp??cialis??es appel??es ADN de t??lom??res. La fonction principale de ces r??gions est de permettre ?? la cellule de se r??pliquer extr??mit??s chromosomiques en utilisant l'enzyme t??lom??rase, comme les enzymes qui reproduisent normalement l'ADN ne peut pas copier l'extr??me 3 'extr??mit??s des chromosomes. Ces plafonds de chromosomes sp??cialis??s aident aussi ?? prot??ger l'ADN se termine, et arr??tent les r??paration de l'ADN syst??mes de la cellule de les traiter comme des dommages ?? corriger. En les cellules humaines, les t??lom??res sont g??n??ralement des longueurs d'ADN simple brin contenant plusieurs milliers de r??p??titions d'une s??quence TTAGGG simple.

Ces s??quences riches en guanine peuvent stabiliser les extr??mit??s des chromosomes par la formation de structures d'ensembles d'unit??s empil??es ?? quatre bases, plut??t que les paires de bases habituelles trouv??es dans d'autres mol??cules d'ADN. Ici, quatre bases guanine former une plaque plate et ces unit??s ?? quatre bases plates ensuite empiler l'une sur l'autre, pour former une ??table La structure G-quadruplex. Ces structures sont stabilis??es par des liaisons hydrog??ne entre les bords des bases et ch??lation d'un ion m??tallique dans le centre de chaque unit?? de quatre bases. D'autres structures peuvent ??galement ??tre form??s, ?? l'ensemble central de quatre bases provenant d'un seul brin soit pli??e autour de la base, ou plusieurs brins parall??les diff??rents, chacun apportant une base pour la structure centrale.
En plus de ces structures empil??es, les t??lom??res forment ??galement de grandes structures en boucle appel??s boucles des t??lom??res, ou T-boucles. Ici, les boucles d'ADN simple brin autour d'une longue cercle stabilis??e par des prot??ines t??lom??res contraignant. A la fin de la boucle T, l'ADN simple brin t??lom??re est maintenu sur une r??gion d'ADN double brin par brin de l'ADN t??lom??re perturber appariement de bases et en double h??lice de l'un des deux brins. Cette structure en triple brin est appel?? une boucle de d??placement ou D-boucle.
 |  |
| Seule branche | Branches multiples |
ADN ramifi??,
Dans l'ADN effilochage se produit lorsque les r??gions non compl??mentaires existent ?? l'extr??mit?? d'un double brin d'ADN compl??mentaire autrement. Cependant, l'ADN ramifi?? peut se produire si un tiers brin d'ADN est introduit et contient des r??gions voisines susceptibles de se hybrider avec les r??gions effiloch??s de la double-brin pr??-existant. Bien que l'exemple le plus simple d'ADN ramifi?? implique seulement trois brins d'ADN, complexes impliquant des brins suppl??mentaires et plusieurs branches sont ??galement possibles. ADN ramifi?? peut ??tre utilis?? dans la nanotechnologie pour construire des formes g??om??triques, voir la section sur les utilisations de la technologie ci-dessous.
Vibration
ADN peut mener basse fr??quence de mouvement collectif observ??e par le Spectroscopie Raman et analys??es avec un mod??le quasi-continuum.
Des modifications chimiques et emballage de l'ADN modifi??
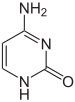 | 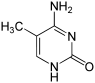 |  |
| cytosine | 5-m??thylcytosine | thymine |
Modifications de base et de l'emballage de l'ADN
L'expression des g??nes est influenc??e par la fa??on dont l'ADN est emball?? dans des chromosomes, dans une structure appel??e chromatine. modifications de base peuvent ??tre impliqu??s dans l'emballage, avec des r??gions qui ont peu ou pas de l'expression des g??nes contenant g??n??ralement des niveaux ??lev??s de m??thylation de des bases cytosine. empaquetage de l'ADN et son influence sur l'expression des g??nes peut ??galement se produire par des modifications covalentes de la prot??ine histone noyau autour duquel est enroul?? l'ADN dans la structure de la chromatine ou bien en remodelant r??alis??e par des complexes de remodelage de la chromatine (voir Remodelage de la chromatine). Il est, en outre, diaphonie entre la m??thylation de l'ADN et de modification d'histone, de sorte qu'ils peuvent affecter de mani??re coordonn??e la chromatine et l'expression g??nique.
Par exemple, la m??thylation de la cytosine, produit 5-m??thylcytosine, ce qui est important pour Inactivation du chromosome X. Le niveau moyen de m??thylation varie entre organismes - le ver Caenorhabditis elegans manque de m??thylation de cytosine, tandis que les vert??br??s ont des niveaux plus ??lev??s, avec un maximum de 1% de leur ADN contenant la 5-m??thylcytosine. Malgr?? l'importance de la 5-m??thylcytosine, il peut d??saminer de quitter une base de thymine, de sorte cytosines m??thyl??s sont particuli??rement sujettes ?? mutations. D'autres modifications comprennent la m??thylation de bases ad??nine chez les bact??ries, la pr??sence de 5-hydroxym??thylcytosine dans le cerveau , et le glycosylation de l'uracile pour produire le "J-base" dans kin??toplastides.
Dommage

L'ADN peut ??tre endommag?? par de nombreuses sortes de mutag??nes, qui changent la s??quence d'ADN. Mutag??nes comprennent agents oxydants, agents et aussi de haute ??nergie alkylants rayonnement ??lectromagn??tique telles que ultraviolet et la lumi??re Les rayons X. Le type d'alt??ration de l'ADN produite d??pend du type de mutag??ne. Par exemple, la lumi??re UV peut endommager l'ADN en produisant dim??res de thymine, qui sont des liaisons transversales entre les bases pyrimidiques. D'autre part, des oxydants tels que les radicaux libres ou peroxyde d'hydrog??ne produisent de multiples formes de dommages, y compris les modifications de base, en particulier de la guanosine, et cassures double brin. Une cellule humaine typique contient environ 150 000 bases qui ont subi des dommages oxydatifs. De ces l??sions oxydatives, les plus dangereux sont cassures double-brin, car ceux-ci sont difficiles ?? r??parer et peuvent produire mutations ponctuelles, insertions et deletions de la s??quence d'ADN, ainsi que translocations chromosomiques. Ces mutations peuvent causer le cancer . En raison des limites inh??rentes aux m??canismes de r??paration d'ADN, si les humains vivaient assez longtemps, ils seraient tous ??ventuellement d??velopper un cancer. dommages d'ADN qui sont d'origine naturelle, en raison de processus cellulaires normaux qui produisent des esp??ces r??actives de l'oxyg??ne, de l'activit?? d'hydrolyse de l'eau cellulaire, etc., apparaissent aussi fr??quemment. Bien que la plupart de ces dommages sont r??par??s, dans une cellule des dommages de l'ADN peut rester malgr?? l'action des processus de r??paration. Ces dommages de l'ADN restants se accumulent avec l'??ge dans les tissus de mammif??res postmitotiques. Cette accumulation semble ??tre une importante cause sous-jacente du vieillissement.
Beaucoup mutag??nes se inscrivent dans l'espace entre deux paires de bases adjacentes, cela se appelle intercalation. La plupart sont intercalateurs mol??cules aromatiques et planes; des exemples comprennent le bromure d'??thidium, acridines, la daunomycine, et doxorubicine. Pour un intercalant pour se adapter entre les paires de base, les bases doivent se s??parer, ce qui fausse les brins d'ADN par d??roulement de la double h??lice. Cela inhibe ?? la fois la transcription et la replication de l'ADN, causant la toxicit?? et des mutations. En cons??quence, les agents intercalants d'ADN peuvent ??tre des agents canc??rig??nes, et dans le cas de la thalidomide, t??ratog??ne. D'autres, comme benzo [a] pyr??ne diol ??poxyde et Formulaire de aflatoxine adduits de l'ADN qui induisent des erreurs dans la r??plication. N??anmoins, en raison de leur aptitude ?? inhiber la transcription et la r??plication de l'ADN, d'autres toxines similaires sont ??galement utilis??s dans la chimioth??rapie pour inhiber croissance rapide cancer cellules.
Fonctions biologiques
ADN se produit g??n??ralement lin??aire chromosomes dans des eucaryotes et des chromosomes circulaires les procaryotes. L'ensemble des chromosomes dans une cellule fait son g??nome; la g??nome humain a environ 3 milliards de paires de bases d'ADN arrang??es en 46 chromosomes. L'information port??e par l'ADN est maintenu dans la s??quence de morceaux d'ADN appel??s g??nes. Transmission de l'information g??n??tique dans les g??nes est r??alis??e par appariement de bases compl??mentaires. Par exemple, dans la transcription, quand une cellule utilise l'information contenue dans un g??ne, la s??quence d'ADN est copi??e dans une s??quence d'ARN compl??mentaire par l'attraction entre l'ADN et les nucleotides d'ARN appropri??s. Habituellement, cette copie d'ARN est ensuite utilis?? pour effectuer une correspondance s??quence de la prot??ine dans un processus appel?? traduction, qui d??pend de la m??me interaction entre les nucl??otides d'ARN. En mode alternative, une cellule peut simplement copier son information g??n??tique dans une r??plication de l'ADN de processus appel??. Les d??tails de ces fonctions sont trait??es dans d'autres articles; ici nous nous concentrons sur les interactions entre l'ADN et d'autres mol??cules qui interviennent dans la fonction du g??nome.
G??nes et des g??nomes
L'ADN g??nomique est ??troitement et ordonn??e emball?? dans le processus appel?? condensation de l'ADN pour se adapter ?? des faibles volumes disponibles de la cellule. Chez les eucaryotes, l'ADN est situ?? dans la noyau de la cellule, ainsi que de petites quantit??s dans les mitochondries et chloroplastes. Chez les procaryotes, l'ADN a lieu au sein d'un corps de forme irr??guli??re dans le cytoplasme appel??e nucl??o??de. L'information g??n??tique dans le g??nome est maintenue ?? l'int??rieur des g??nes, et l'ensemble complet de ces informations dans un organisme est appel?? le g??notype. Un g??ne est une unit?? de h??r??dit?? et est une r??gion d'ADN qui influence une caract??ristique particuli??re dans un organisme. Les g??nes contiennent une cadre de lecture ouvert qui peut ??tre transcrit, ainsi que des s??quences r??gulatrices telles que promoteurs et des activateurs qui contr??lent la transcription du cadre de lecture ouvert.
Dans de nombreuses esp??ces , seule une petite fraction de la s??quence totale de la g??nome code pour la prot??ine. Par exemple, seulement environ 1,5% du g??nome humain est constitu?? de prot??ines de codage exons, dont plus de 50% de l'ADN humain comprenant des non codante s??quences r??p??titives. Les raisons de la pr??sence de beaucoup ADN non codant dans les g??nomes eucaryotes et les diff??rences extraordinaires la taille du g??nome, ou C-valeur, parmi les esp??ces repr??sentent un casse-t??te depuis longtemps connu comme le " Paradoxe de la valeur C ". Cependant, certaines s??quences d'ADN qui ne ont pas la prot??ine de code peuvent encore encoder fonctionnelle non codante des mol??cules d'ARN, qui sont impliqu??s dans la r??gulation de l'expression g??nique.

Certaines s??quences d'ADN non codantes jouent un r??le structurel dans les chromosomes. Les t??lom??res et centrom??res contiennent typiquement quelques g??nes, mais sont importantes pour la fonction et la stabilit?? des chromosomes. Un formulaire d'ADN non codant abondante chez les humains sont pseudog??nes, qui sont des copies de g??nes qui ont ??t?? d??sactiv??s par mutation. Ces s??quences sont habituellement seulement mol??culaires fossiles , m??me si elles peuvent parfois servir de mati??re premi??re pour la g??n??tique comme la cr??ation de nouveaux g??nes dans le processus de la duplication de g??nes et divergence.
Transcription et traduction
Un g??ne est une s??quence d'ADN qui contient l'information g??n??tique et peuvent influencer le ph??notype d'un organisme. Au sein d'un g??ne, la s??quence de bases le long d'un brin d'ADN d??finit un s??quence d'ARN messager, qui d??finit alors une ou plusieurs des s??quences de prot??ines. La relation entre les s??quences nucl??otidiques des g??nes et les acides amin??s des s??quences de prot??ines est d??termin??e par les r??gles de traduction, connus collectivement sous le code g??n??tique . Le code g??n??tique est constitu?? de trois lettres appel??s codons les "mots de form??s ?? partir d'une s??quence de trois nucl??otides (par exemple la loi, l'ACG, TTT).
Dans la transcription, des codons d'un g??ne sont copi??s en ARN messager par ARN polym??rase. Cette copie de l'ARN est ensuite d??cod?? par un ribosome qui lit la s??quence d'ARN par appariement de bases de l'ARN messager ARN de transfert, qui porte les acides amin??s. Comme il ya quatre bases dans des combinaisons de 3 lettres, il ya 64 codons possibles (  combinaisons). Ceux-ci codent pour la vingt acides amin??s standards, des acides amin??s donnant plus plus d'un codon possible. Il ya aussi trois ??stop?? ou ??codons non-sens?? signifiant la fin de la r??gion codante; ce sont les TAA, TGA et codons TAG.
combinaisons). Ceux-ci codent pour la vingt acides amin??s standards, des acides amin??s donnant plus plus d'un codon possible. Il ya aussi trois ??stop?? ou ??codons non-sens?? signifiant la fin de la r??gion codante; ce sont les TAA, TGA et codons TAG.

Replication
La division cellulaire est essentielle ?? l'organisme de se d??velopper, mais, quand une cellule se divise, elle doit se r??pliquer de l'ADN dans le g??nome de sorte que les deux cellules filles ont la m??me information g??n??tique que leur parent. La structure double brin de l'ADN fournit un m??canisme simple pour r??plication de l'ADN. Ici, les deux brins sont s??par??s et puis chaque brin de s??quence d'ADN compl??mentaire est recr???? par un enzyme appel??e ADN polym??rase. Cette enzyme permet le brin compl??mentaire en trouvant la base correcte par appariement de bases compl??mentaires, et la liaison de-la sur le brin d'origine. Comme les ADN polymerases peuvent se ??tendre seulement un brin d'ADN en 5 '?? 3', diff??rents m??canismes sont utilis??s pour copier les brins antiparall??les de la double h??lice. De cette fa??on, la base sur les anciens pr??ceptes de brins dont la base se affiche sur le nouveau brin, et la cellule se retrouve avec une copie parfaite de son ADN.
Interactions avec les prot??ines
Toutes les fonctions de l'ADN d??pendent des interactions avec les prot??ines. Ces interactions prot??iques peuvent ??tre non sp??cifique, ou la prot??ine peut se lier sp??cifiquement ?? une s??quence d'ADN unique. Les enzymes peuvent ??galement se lier ?? l'ADN et de ceux-ci, les polymerases qui copient la s??quence de bases de l'ADN dans la transcription et la r??plication de l'ADN sont particuli??rement importants.
prot??ines de liaison ?? l'ADN
 |
Prot??ines structurelles qui lient l'ADN sont des exemples bien compris des interactions non-sp??cifiques ADN-prot??ines. Dans chromosomes, l'ADN est maintenu en complexes avec des prot??ines structurelles. Ces prot??ines organisent l'ADN dans une structure compacte appel?? chromatine. Chez les eucaryotes cette structure de liaison d'ADN consiste en un complexe de petites prot??ines basiques appel??e histones, tandis que chez les procaryotes plusieurs types de prot??ines sont impliqu??es. Les histones forment un complexe en forme de disque appel?? nucl??osome, qui contient deux tours complets de l'ADN double brin enroul?? autour de sa surface. Ces interactions non sp??cifiques sont form??s par des r??sidus de base dans la fabrication histones liaisons ioniques ?? l'acide le sucre-phosphate ??pine dorsale de l'ADN, et sont donc en grande partie ind??pendante de la s??quence de base. Des modifications chimiques de ces r??sidus basiques d'acides amin??s comprennent la m??thylation, la phosphorylation et ac??tylation. Ces modifications chimiques modifient la force de l'interaction entre l'ADN et des histones, ce qui rend l'ADN plus ou moins accessible ?? facteurs de transcription et la modification du taux de transcription. D'autres prot??ines de liaison ?? l'ADN non-sp??cifiques dans la chromatine comprennent les prot??ines du groupe de haute mobilit??, qui se lient ?? l'ADN courb?? ou d??form??. Ces prot??ines sont importantes en flexion des tableaux de nucl??osomes et les disposer dans les plus grandes structures qui composent les chromosomes.
Un groupe distinct de prot??ines de liaison ?? l'ADN sont les prot??ines de liaison ?? l'ADN qui se lient sp??cifiquement l'ADN simple brin. Chez l'homme, la replication la prot??ine A est le membre le plus entendu de cette famille et est utilis?? dans des proc??d??s o?? la double h??lice est s??par??, y compris la r??plication de l'ADN, la recombinaison et la r??paration de l'ADN. Ces prot??ines de liaison semblent stabiliser l'ADN simple brin et le prot??ger de la formation souches boucles ou d'??tre d??grad?? par nucl??ases.

En revanche, d'autres prot??ines ont ??volu?? pour se lier ?? des s??quences d'ADN particuli??res. Le plus intensivement ??tudi?? d'entre eux sont diff??rents facteurs de transcription, qui sont des prot??ines qui r??gulent la transcription. Chaque facteur de transcription se lie ?? un ensemble particulier de s??quences d'ADN et active ou inhibe la transcription de g??nes qui ont ces s??quences ?? proximit?? de leurs promoteurs. Les facteurs de transcription font de deux fa??ons. Tout d'abord, ils peuvent se lier la polymerase de transcription responsable de l'ARN, soit directement, soit par d'autres m??diateurs prot??iques; cette localise la polymerase au promoteur et lui permet de lancer la transcription. En variante, des facteurs de transcription peuvent se lier enzymes qui modifient les histones au promoteur. Ceci modifie l'accessibilité de la matrice d'ADN à la polymerase.
Étant donné que ces cibles d'ADN peuvent se produire tout au long du génome d'un organisme, des changements dans l'activité d'un type de facteur de transcription peuvent influer sur des milliers de gènes. Par conséquent, ces protéines sont souvent les cibles des processus de transduction de signaux qui contrôlent les réponses aux changements de l'environnement ou la différenciation cellulaire et le développement. La spécificité des interactions de ces facteurs de transcription à l'ADN proviennent des protéines faisant multiples contacts avec les bords des bases de l'ADN, ce qui leur permet de "lire" la séquence d'ADN. La plupart de ces bases-interactions sont faites dans le grand sillon, où les bases sont plus accessibles.

les enzymes modifiant l'ADN
Nucléases et ligases
Les nucléases sont des enzymes qui coupent les brins d'ADN par la catalyse de la hydrolyse des liaisons phosphodiester. Nucleases qui hydrolysent les nucléotides de l'extrémité de brins d'ADN sont appelées exonucléases, tandis que les endonucléases coupent à l'intérieur des brins. Les nucleases plus souvent utilisés dans la biologie moléculaire sont les endonucléases de restriction, qui coupent l'ADN au niveau de séquences spécifiques. Par exemple, l'enzyme EcoRV montré à la gauche reconnaît la séquence de base 6-5'-GATATC-3 'et effectue une coupe à la ligne verticale. Dans la nature, ces enzymes protéger les bactéries contre l'infection par le phage par digestion de l'ADN de phage lorsqu'il entre dans la cellule bactérienne, en qualité d'une partie du système de modification de restriction. Dans la technologie, ces nucléases spécifiques de séquence sont utilisés dans le clonage moléculaire et l'empreinte génétique.
Enzymes appel??s ligases d'ADN peuvent rejoindre brins d'ADN coupés ou cassés. Ligases sont particulièrement importantes dans la réplication de l'ADN du brin tardif, où ils se réunissent les courts segments d'ADN produite à la fourche de réplication dans une copie complète de la matrice d'ADN. Ils sont également utilisés dans la réparation de l'ADN et la recombinaison génétique.
Topoisomerases et hélicases
Topoisomérases sont des enzymes à la fois avec la nucléase et l'activité de ligase. Ces protéines changent la quantité de superenroulement dans l'ADN. Certaines de ces enzymes fonctionnent en coupant l'hélice d'ADN et une section permettant de tourner, ce qui réduit son niveau de surenroulement; l'enzyme scelle la cassure de l'ADN. D'autres types de ces enzymes sont capables de couper une hélice d'ADN et ensuite passer un second brin d'ADN à travers cette pause, avant de rejoindre l'hélice. Les topoisomérases sont nécessaires pour de nombreux procédés impliquant l'ADN, tels que la replication de l'ADN et la transcription.
Les hélicases sont des protéines qui sont un type de moteur moléculaire. Ils utilisent l'énergie chimique en triphosphates nucléosidiques, principalement ATP , pour rompre les liaisons hydrogène entre les bases et détendez-vous la double hélice d'ADN en brins simples. Ces enzymes sont essentielles pour la plupart des processus où les enzymes ont besoin pour accéder aux bases de l'ADN.
Polymérases
Polymérases sont des enzymes qui synthétisent chaînes polynucléotidiques de nucléosides triphosphates. La séquence de leurs produits sont des copies de chaînes-qui polynucléotidiques existants sont appelés modèles . Ces enzymes fonctionnent en ajoutant des nucleotides sur la 3 ' groupe hydroxyle du nucleotide précédent dans un brin d'ADN. En conséquence, toutes les polymerases travaillent dans une direction 5 'à 3'. Dans le site actif de ces enzymes, les triphosphates de nucléoside paires de bases entrants vers le modèle: ceci permet de synthétiser des polymerases avec précision le brin complémentaire de la matrice. Polymerases sont classées selon le type de modèle qu'ils utilisent.
Dans la réplication de l'ADN, un ADN-dépendante de l'ADN polymerase effectue une copie d'une séquence d'ADN. Précision est vital dans ce processus, de sorte que bon nombre de ces polymérases avoir une activité de relecture. Ici, la polymérase reconnaît les erreurs occasionnelles dans la réaction de synthèse par le manque d'appariement de bases entre les nucléotides dépareillées. Si une incohérence est détectée, un 3 'à 5' exonucléase est activé et la base incorrect enlevé. Dans la plupart des organismes, fonction des polymérases d'ADN dans un grand complexe appelé replisome qui contient plusieurs sous-unités accessoires, tels que la pince de l'ADN ou les hélicases.
ADN polymerases ARN-dépendantes sont une classe spécialisée des polymerases qui copient la séquence d'un brin d'ARN en ADN. Ils comprennent transcriptase inverse, qui est un viral enzyme impliquée dans l'infection des cellules par des retrovirus, et la télomérase, qui est nécessaire pour la réplication des télomères . La télomérase est une polymérase inhabituel parce qu'il contient sa propre matrice d'ARN dans le cadre de sa structure.
La transcription est effectuée par un ADN-dépendante de l'ARN polymerase qui copie la séquence d'un brin d'ADN en ARN. Pour commencer la transcription d'un gène, l'ARN polymerase se lie à une séquence d'ADN appelée un promoteur et sépare les brins d'ADN. Il a ensuite copies de la séquence de gène dans un transcrit d'ARN messager jusqu'à ce qu'il atteigne une région d'ADN appelée terminateur, où il arrête et se détache de l'ADN. Comme dans le cas des ADN-polymérases ADN-dépendantes, les droits de l'ARN polymerase II, l'enzyme qui transcrit la plupart des gènes dans le génome humain, fonctionne comme une partie d'un grand complexe de protéine de sous-unités régulatrices et accessoires multiples.
La recombinaison génétique
 |
 |

Une hélice d'ADN n'a généralement pas d'interaction avec d'autres segments d'ADN, dans des cellules humaines et les différents chromosomes même occupent des zones séparées dans le noyau appelé «territoires chromosomiques". Cette séparation physique des chromosomes différents est important pour la capacité de l'ADN à fonctionner en tant que référentiel stable pour information, comme l'un des quelques fois au cours de chromosomes interagissent est croisement chromosomique quand ils se recombinent. Chromosomique croisement est quand deux hélices d'ADN se cassent, échangent une section puis rejoignent.
Recombinaison permet chromosomes d'échanger des informations génétiques et produit de nouvelles combinaisons de gènes, ce qui augmente l'efficacité de la sélection naturelle et peut être important dans l'évolution rapide des nouvelles protéines. La recombinaison génétique peut également être impliqué dans la réparation d'ADN, en particulier dans la réponse de la cellule de cassures double-brin.
La forme la plus commune de croisement chromosomique est recombinaison homologue, où les deux chromosomes impliqués part séquences très similaires. Recombinaison non homologue peut être dommageable pour les cellules, car il peut produire des translocations chromosomiques et anomalies génétiques. La réaction de recombinaison est catalysée par des enzymes connues sous le nom recombinases, tels que RAD51. La première étape de recombinaison est une coupure double-brin causé soit par une endonucléase ou des dommages à l'ADN. Une série d'étapes catalysées en partie par la recombinase conduit alors à la jonction des deux hélices par au moins une jonction de Holliday, dans lequel un segment d'un brin unique de chaque hélice est recuit au brin complémentaire de l'autre hélice. La jonction de Holliday est une structure tétraédrique de jonction qui peut être déplacé le long de la paire de chromosomes, une permutation de l'autre brin. La réaction de recombinaison est arrêtée par coupure de la jonction et re-ligature de l'ADN connues.
??volution
L'ADN contient l'information génétique qui permet toutes les choses de la vie moderne de fonctionner, grandissent et se reproduisent. Cependant, on ne sait pas combien de temps dans les 4 milliards d'années l'histoire de l'ADN de la vie a assumé cette fonction, comme il a été proposé que les premières formes de vie peuvent avoir utilisé l'ARN comme matériel génétique. ARN peut avoir agi comme la partie centrale du début métabolisme cellulaire comme il peut à la fois transmettre des informations génétiques et de réaliser la catalyse dans le cadre de ribozymes. Cet ancien monde de l'ARN où acide nucléique aurait été utilisé à la fois pour la catalyse et de la génétique peut avoir influencé l' évolution du code génétique actuel, basé sur quatre bases nucléotidiques. Cela se produit, étant donné que le nombre de bases différentes dans un tel organisme est un compromis entre un petit nombre de bases en augmentant la précision de réplication et un grand nombre de bases d'accroître l'efficacité catalytique des ribozymes.
Cependant, il n'y a pas de preuve directe de systèmes génétiques anciennes, comme la récupération de l'ADN de la plupart des fossiles est impossible. Ceci est parce que l'ADN survit dans l'environnement pendant moins d'un million d'années, et se dégrade lentement dans de courts fragments en solution. Revendications pour l'ADN âgés ont été faites, notamment un rapport de l'isolement d'une bactérie viable à partir d'un cristal de sel vieux de 250 millions d'années, mais ces revendications sont controversées.
Le 8 Août 2011, un rapport, sur la base dela NASAétudes avecmétéorites trouvées surTerre, a été publié suggérant blocs constitutifs de l'ADN ( ad??nine, guanine et connexesmolécules organiques) peuvent avoir été formé dans extraterrestrially cosmos.
Utilise la technologie
Ingénierie génétique
Des méthodes ont été développées pour purifier l'ADN à partir d'organismes tels que l'extraction au phénol-chloroforme, et à manipuler en laboratoire, telles que les digestions de restriction et la réaction en chaîne de la polymérase . Moderne biologie et biochimie font un usage intensif de ces techniques de la technologie de l'ADN recombinant. ADN recombinant est une séquence d'ADN artificielle, qui a été assemblé à partir d'autres séquences d'ADN. Ils peuvent être transformés en les organismes sous la forme de plasmides ou dans le format approprié, par utilisation d'un vecteur viral. Le organismes génétiquement modifiés produits peuvent être utilisés pour produire des produits de recombinaison tels que des protéines , utilisés dans la recherche médicale, ou être cultivées en agriculture .
Forensics
Les experts judiciaires peuvent utiliser l'ADN dans le sang , le sperme, la peau, la salive ou les cheveux trouvés à une scène de crime pour identifier un ADN correspondant d'un individu, comme un agresseur. Ce processus est formellement appelé profilage ADN, mais peut également être appelé " empreinte génétique ". Dans profilage ADN, les longueurs des sections variables d'ADN répétitif, comme de courtes répétitions en tandem et minisatellites, sont comparées entre les gens. Cette méthode est habituellement une technique extrêmement fiable pour identifier un ADN correspondant. Cependant, l'identification peut être compliqué si la scène est contaminée avec de l'ADN de plusieurs personnes. Profilage ADN a été développé en 1984 par le généticien britannique Sir Alec Jeffreys, et d'abord utilisé dans la science médico-légale pour condamner Colin Pitchfork en 1988 meurtres cas Enderby.
Le développement de la science médico-légale, et la capacité d'obtenir maintenant adaptation génétique sur des échantillons infimes de sang, la peau, la salive ou les cheveux a conduit à un réexamen d'un certain nombre de cas. La preuve peut maintenant être découvert cela n'a pas été scientifiquement possible au moment de l'examen initial. Combiné avec la suppression de la loi de la double incrimination dans certains endroits, ce qui peut permettre à des cas à être rouvertes où des essais précédents ont échoué à produire une preuve suffisante pour convaincre un jury. Les personnes accusées de crimes graves peuvent être tenus de fournir un échantillon d'ADN pour fins d'appariement. La défense la plus évidente pour les matchs d'ADN obtenus médico-légal est de prétendre que la contamination croisée de la preuve a eu lieu. Cela a abouti à des procédures de manipulation strictes méticuleux avec de nouveaux cas de crimes graves. Profilage ADN est également utilisé pour identifier les victimes des incidents de pertes massives. Ainsi que d'identifier positivement les corps ou parties de corps dans des accidents graves, profilage ADN est utilisé avec succès pour identifier les victimes individuelles dans des tombes de guerre de masse - correspondant aux membres de la famille.
Bioinformatique
Bioinformatique implique la manipulation, la recherche et l'extraction de données de données biologiques, et cela comprend les données de séquences d'ADN. Le développement des techniques pour stocker et rechercher des séquences d'ADN ont conduit à largement avancées appliquées à l'informatique , en particulier les algorithmes de recherche de chaînes, apprentissage de la machine et la théorie de base de données. Chaîne de recherche ou algorithmes, qui trouvent une occurrence d'une séquence de lettres à l'intérieur d'une plus grande séquence de lettres correspondant, ont été développés pour rechercher des séquences spécifiques de nucléotides. La séquence d'ADN peut être aligné avec d'autres séquences d'ADN pour identifier les séquences homologues spécifiques et localiser les mutations qui les rendent distinct. Ces techniques, en particulier l'alignement de séquences multiples , sont utilisés dans l'étude des relations phylogénétiques et la fonction des protéines. Les ensembles de données représentant la valeur de séquences d'ADN, telles que celles produites par le de génomes entiers Projet du génome humain, sont difficiles à utiliser sans les annotations qui identifient les emplacements des gènes et des éléments de régulation sur chaque chromosome. Les régions de séquence d'ADN qui ont les configurations caractéristiques associées aux gènes en protéines ou d'ARN codant peuvent être identifiés par des Gènes trouver des algorithmes qui permettent aux chercheurs de prédire la présence de certains produits de gènes et de leurs fonctions possibles dans un organisme avant même qu'elles ont été isolées expérimentalement. Génomes entiers peuvent également être comparés, ce qui peut faire la lumière sur l'histoire évolutive de l'organisme et notamment permettre l'examen des événements évolutifs complexes.
Nanotechnologie de l'ADN

Nanotechnologie de l'ADN utilise les uniques propriétés de reconnaissance moléculaire de l'ADN et d'autres acides nucléiques pour créer des complexes d'ADN ramifiés auto-assemblage avec des propriétés utiles. L'ADN est donc utilisé en tant que matériau de structure plutôt que comme un support d'informations biologiques. Cela a conduit à la création de réseaux périodiques à deux dimensions (deux carreaux à base ainsi que l'aide de la " origami ADN "de la méthode) ainsi que des structures en trois dimensions dans les formes de polyèdres . dispositifs nanomécaniques et algorithmique auto-assemblage ont également été démontrée, et ces structures d'ADN ont été utilisées à le modèle de l'agencement d'autres molécules telles que des nanoparticules d'or et des protéines de streptavidine.
Histoire et anthropologie
Parce que l'ADN recueille mutations au fil du temps, qui sont ensuite hérité, il contient des informations historiques, et, en comparant les séquences d'ADN, les généticiens peuvent déduire l'histoire évolutive des organismes, leur phylog??nie. Ce domaine de la phylogénétique est un outil puissant dans la biologie évolutive . Si les séquences d'ADN d'une espèce sont comparées, les généticiens des populations peuvent apprendre l'histoire des populations particulières. Ceci peut être utilisé dans des études allant de la génétique écologiques anthropologie ; Par exemple, la preuve d'ADN est utilisée pour tenter d'identifier les dix tribus perdues d'Israël.
L'ADN a également été utilisé pour examiner les relations familiales modernes, tels que l'établissement de relations familiales entre les descendants de Sally Hemings et Thomas Jefferson . Cet usage est étroitement liée à l'utilisation de l'ADN dans les enquêtes criminelles détaillées ci-dessus. En effet, certaines enquêtes criminelles ont été résolus lorsque l'ADN à partir de scènes de crime a égalé parents de l'individu coupable.
stockage de l'information
Dans un article publié dans Nature en Janvier 2013, des scientifiques de l' Institut de bioinformatique et européennes Agilent Technologies a proposé un mécanisme pour utiliser la capacité de l'ADN pour coder l'information comme un moyen de stockage de données numériques. Le groupe était capable d'encoder 739 kilo-octets de données dans le code de l'ADN, la synthèse de l'ADN réelle, puis séquencer l'ADN et de décoder les informations de retour à sa forme originale, avec une précision rapporté 100%. L'information codée composée de fichiers texte et des fichiers audio. Une expérience préalable a été publié en Août 2012. Elle a été menée par des chercheurs de l'Université Harvard, où le texte d'un livre 54 000 mots a été codé dans l'ADN.
Histoire de la recherche de l'ADN

L'ADN a été isolé pour la première par le suisse médecin Friedrich Miescher qui, en 1869, a découvert une substance microscopique dans le pus des bandages chirurgicaux jetés. Comme il résidait dans les noyaux des cellules, il l'a appelé "nucléine". En 1878, Albrecht Kossel isolé le composant non protéique de "nucléine", l'acide nucléique, et plus tard isolé de ses cinq principaux nucléobases. En 1919, Phoebus Levene identifié l'unité de base, le sucre et le phosphate nucléotidique. Levene a suggéré que l'ADN est composée d'une chaîne d'unités nucléotidiques reliées entre elles par l'intermédiaire des groupes phosphate. Cependant, Levene pensait la chaîne était court et les bases répétée dans un ordre fixe. En 1937, William Astbury a produit les premiers modèles de diffraction des rayons X qui ont montré que l'ADN avait une structure régulière.
En 1927, Nikolai Koltsov a proposé que les traits héréditaires seraient hérités via une «molécule héréditaire géant» composée de «deux brins de miroir qui se répliquer d'une manière semi-conservatrice en utilisant chaque brin comme un modèle". En 1928, Frederick Griffith a découvert que les traits de la "douceur" forme de pneumocoque pourrait être transférée à la forme "brute" des mêmes bactéries en mélangeant bactéries tuées «lisses» avec le formulaire en direct "rough". Ce système a fourni la première suggestion clair que l'ADN porte l'information génétique-le Avery-MacLeod-McCarty expérience-quand Oswald Avery, le long avec des collègues Colin MacLeod et Maclyn McCarty, ADN identifié comme le principe de la transformation du rôle de 1943. ADN dans l'hérédité a été confirmée dans 1952, Alfred Hershey et Martha Chase dans le Hershey-Chase expérience a montré que l'ADN est le matériel génétique du phage T2.
En 1953,James D. WatsonetFrancis Crickont suggéré ce qui est maintenant accepté comme le premier modèle à double hélice correcte dela structure de l'ADN dans la revue Nature.Leur double hélice, le modèle moléculaire de l'ADN a ensuite été basé sur une seuleimage de diffraction des rayons X ( étiquetés comme «Photo 51 ") prise par Rosalind Franklin et Raymond Gosling mai 1952, ainsi que les informations que les bases de l'ADN sont jumelés - également obtenus grâce à des communications privées deErwin Chargaff dans les années précédentes.règles de chargaff joué un rôle très important dans l'établissement à double configurations hélice pour l'ADN-B, ainsi que l'ADN-A.
Des preuves expérimentales soutenant le Watson et Crick modèle a été publié dans une série de cinq articles dans le même numéro de Nature . Parmi ceux-ci, le document de Franklin et Gosling était la première publication de leurs propres données de diffraction des rayons X et la méthode d'analyse originale que partiellement pris en charge le modèle de Watson et Crick; cette question contenait également un article sur la structure de l'ADN par Maurice Wilkins et deux de ses collègues, dont l'analyse et in vivo modèles B-ADN rayons X ont également soutenu la présence in vivo des configurations à double hélice d'ADN tel que proposé par Crick et Watson leur modèle moléculaire à double hélice de l'ADN dans les deux pages précédentes de la nature . En 1962, après la mort de Franklin, Watson, Crick et Wilkins reçu conjointement le prix Nobel de physiologie ou médecine. Prix ??????Nobel ont été attribués seulement aux bénéficiaires vivant à l'époque. Un débat se poursuit au sujet de qui devrait recevoir un crédit pour la découverte.
Dans une présentation influente en 1957, Crick aménagé le dogme central de la biologie moléculaire, qui prédit la relation entre l'ADN, l'ARN et les protéines, et articulé le «adaptateur hypothèse". La confirmation définitive du mécanisme de réplication qui a été impliqué par la structure en double hélice suivi en 1958 par l' expérience Meselson-Stahl. D'autres travaux par Crick et ses collègues ont montré que le code génétique a été basé sur des triplets non-chevauchement des bases, appelés codons, permettant Har Gobind Khorana, Robert W. Holley et Marshall Nirenberg Warren à déchiffrer le code génétique. Ces résultats représentent la naissance de biologie mol??culaire.

