
r??action en cha??ne de la polym??rase
Saviez-vous ...
SOS croit que l'??ducation donne une meilleure chance dans la vie des enfants dans le monde en d??veloppement aussi. M??res SOS chaque regard apr??s une une famille d'enfants parrain??s .
La r??action en cha??ne par polymerase (PCR) est une technique largement utilis??e dans biologie mol??culaire. Il tire son nom de l'un de ses ??l??ments cl??s, un ADN polym??rase utilis??e pour amplifier un fragment de l'ADN par in vitro enzymatique r??plication. Comme progresse PCR, l'ADN ainsi produit est lui-m??me utilis?? comme matrice pour la r??plication. Cela d??clenche une r??action en cha??ne dans laquelle la matrice d'ADN est exponentielle amplifi??. Avec la PCR, il est possible d'amplifier une seule ou quelques copies d'un fragment d'ADN dans plusieurs ordres de grandeur, des millions de g??n??ration ou de plusieurs copies du morceau d'ADN. La PCR peut ??tre largement modifi?? pour ex??cuter un large ??ventail de manipulations g??n??tiques.
La quasi-totalit?? des applications de PCR utilisent une ADN polymerase thermostable, telle que Taq polym??rase, une enzyme isol??e ?? l'origine ?? partir de la bact??rie Thermus aquaticus. Cet ADN polym??rase enzymatique assemble un nouveau brin d'ADN ?? partir de blocs de construction ADN, le nucleotides, en utilisant l'ADN simple brin comme matrice et oligonucleotides d'ADN (??galement appel??s des amorces d'ADN) n??cessaires pour l'initiation de la synth??se d'ADN. La grande majorit?? des m??thodes PCR utilisation cyclage thermique, ce est ?? dire, alternativement chauffer et refroidir l'??chantillon de PCR ?? une s??rie d??finie d'??tapes de temp??rature. Ces ??tapes thermiques de cyclisme est n??cessaire de s??parer physiquement les brins (?? haute temp??rature) dans une double h??lice d'ADN ( ADN fusion) utilis?? comme mod??le lors de la synth??se d'ADN (?? des temp??ratures inf??rieures) par l'ADN polym??rase pour amplifier s??lectivement l'ADN cible. La s??lectivit?? de PCR r??sultant de l'utilisation de des amorces qui sont compl??mentaire de la r??gion d'ADN cibl??e pour l'amplification dans des conditions thermiques sp??cifiques de cyclisme.
D??velopp?? en 1983 par Kary Mullis, PCR est maintenant une technique courante et souvent indispensable utilis?? dans les laboratoires de recherche m??dicale et biologique pour une vari??t?? d'applications. Il se agit notamment clonage de l'ADN pour bas?? sur le s??quen??age d'ADN, phylog??nie, ou analyse fonctionnelle des g??nes; le diagnostic de maladies h??r??ditaires; l'identification de empreintes g??n??tiques (utilis??s dans sciences m??dico-l??gales et les tests de paternit??); et la d??tection et le diagnostic de maladies infectieuses . En 1993 Mullis a remport?? le prix Nobel de chimie pour ses travaux sur la PCR.
Principes et la proc??dure PCR


La PCR est utilis??e pour amplifier des r??gions sp??cifiques d'un brin d'ADN (l'ADN cible). Il peut se agir d'un seul g??ne, une partie d'un g??ne ou une s??quence non codante. La plupart des m??thodes de PCR amplifient g??n??ralement des fragments d'ADN allant jusqu'?? 10 paires de bases (kb kilo), bien que certaines techniques permettent une amplification de fragments jusqu'?? 40 kb.
Une PCR base mis en place n??cessite plusieurs composants et r??actifs. Ces composants incluent:
- matrice d'ADN qui contient la r??gion d'ADN (cible) ?? amplifier.
- Deux amorces, qui sont compl??mentaires des r??gions d'ADN ?? la 5 '(cinq prime) ou 3 '(de trois prime) extr??mit??s de la r??gion d'ADN.
- Un ADN polymerase telle que La polymerase Taq ou une autre polymerase d'ADN avec une temp??rature optimale autour de 70 ?? C.
- D??soxynucl??osides triphosphates (dNTP; aussi tr??s souvent et ?? tort appel??s d??soxynucl??otide triphosphates), les blocs de construction ?? partir de laquelle les ADN polym??rases synth??tise un nouveau brin d'ADN.
- Solution tampon, fournissant un environnement chimique appropri?? pour une activit?? optimale et la stabilit?? de l'ADN polym??rase.
- Bivalents cations , magn??sium ou de mangan??se ions; g??n??ralement on utilise Mg 2+, Mn 2+, mais peut ??tre utilis?? pour la mutagen??se m??di??e par PCR de l'ADN, comme la concentration plus ??lev??e en Mn 2+ augmente le taux d'erreur au cours de la synth??se d'ADN
- Cation monovalent potassium ions.
La PCR est g??n??ralement effectu??e dans un volume r??actionnel de 10 ?? 200 ul dans de petits tubes de r??action (0,2 ?? 0,5 ml volumes) dans un thermocycleur. Le cycleur thermique chauffe et refroidit les tubes de r??action pour atteindre les temp??ratures n??cessaires ?? chaque ??tape de la r??action (voir ci-dessous). De nombreux thermocycleurs modernes font usage de la Effet Peltier qui permet le chauffage et le refroidissement du bloc de maintien des tubes de PCR en inversant simplement le courant ??lectrique. Tubes de r??action ?? parois minces permettent conductivit?? thermique favorable pour permettre l'??quilibrage thermique rapide. La plupart des thermocycleurs ont couvercles chauffants pour ??viter la condensation au sommet du tube de r??action. Thermocycleurs ??g??es d??pourvues de couvercle chauffant n??cessitent une couche d'huile sur le dessus du m??lange de r??action ou une boule de cire ?? l'int??rieur du tube.
Proc??dure
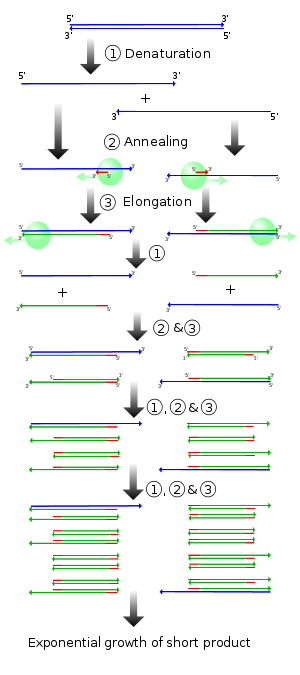
La PCR est g??n??ralement constitu?? d'une s??rie de 20 ?? 40 les variations de temp??rature r??p??t??es appel??es cycles; chaque cycle se compose g??n??ralement de deux ?? trois niveaux de temp??rature distincts. Le plus souvent, la PCR est effectu??e avec des cycles ayant trois paliers de temp??rature (fig. 2). Le v??lo est souvent pr??c??d??e par une ??tape de temp??rature unique (appel?? de maintien) ?? une temp??rature ??lev??e (> 90 ?? C), et suivi par une attente ?? la fin de l'extension du produit final ou une br??ve stockage. Les temp??ratures utilis??es et de la dur??e pendant laquelle ils sont appliqu??s dans chaque cycle d??pendent d'une vari??t?? de param??tres. Ceux-ci comprennent l'enzyme utilis??e pour la synth??se de l'ADN, la concentration des ions divalents et des dNTP ?? la r??action, et la temp??rature de fusion ( Tm) des amorces.
- l'??tape d'initialisation: Cette ??tape consiste ?? chauffer la r??action ?? une temp??rature de 94-96 ?? C (ou 98 ?? C si polymerases thermostables sont extr??mement employ??es), qui est maintenu ?? 1-9 minutes. Il est seulement n??cessaire pour les ADN polym??rases qui n??cessitent l'activation de la chaleur par PCR de d??marrage ?? chaud .
- ??tape de d??naturation: Cette ??tape est le premier ??v??nement cycliste r??gulier et consiste ?? chauffer la r??action ?? 94-98 ?? C pendant 20-30 secondes. Il provoque la fusion de la matrice d'ADN et des amorces de rupture des liaisons hydrog??ne entre les bases compl??mentaires des brins d'ADN, ce qui donne des simples brins d'ADN.
- l'??tape de recuit: La temp??rature de r??action est abaiss??e ?? 50-65 ?? C pendant 20 ?? 40 secondes permettant hybridation des amorces ?? la matrice d'ADN simple brin. Typiquement, la temp??rature de recuit est d'environ 3 ?? 5 degr??s Celsius en dessous de la Tm des amorces utilis??es. Liaisons hydrog??ne ADN-ADN stable ne se forment lorsque la s??quence de l'amorce correspond de tr??s pr??s la s??quence mod??le. La polymerase se lie ?? l'hybride amorce-matrice et commence la synth??se d'ADN.
- Extension / allongement ??tape: La temp??rature ?? cette ??tape d??pend de la polym??rase d'ADN utilis??e; Taq polymerase a son optimum temp??rature de l'activit?? ?? 75 ?? 80 ?? C, et g??n??ralement ?? une temp??rature de 72 ?? C est utilis?? avec cette enzyme. A ce stade de l'ADN polym??rase synth??tise un nouveau brin d'ADN compl??mentaire au brin matrice d'ADN par addition de dNTP qui sont compl??mentaires de la matrice en 5 '?? 3', 5'-condensation de la groupe phosphate des dNTP avec la 3' un groupe hydroxyle ?? l'extr??mit?? de la naissante (extension) de brin d'ADN. Le temps d'extension d??pend ?? la fois l'ADN polym??rase utilis??e et de la longueur du fragment d'ADN ?? amplifier. En r??gle empirique, ?? sa temp??rature optimale, l'ADN polym??rase va polym??riser un millier de bases par minute. Dans des conditions optimales, ?? savoir, se il n'y a pas de limitations dues ?? la limitation des substrats ou des r??actifs, ?? chaque ??tape d'extension, la quantit?? d'ADN cible est doubl??e, ce qui conduit ?? exponentielle (g??om??trique) l'amplification du fragment d'ADN sp??cifique.
- Allongement final: Cette ??tape unique est parfois effectu??e ?? une temp??rature de 70-74 ?? C pendant 5 ?? 15 minutes apr??s le dernier cycle de PCR pour se assurer que tout l'ADN simple brin restant est compl??tement ??tendu.
- Attente final: Cette ??tape ?? 4-15 ?? C pour une dur??e ind??termin??e peut ??tre utilis?? pour le stockage ?? court terme de la r??action.
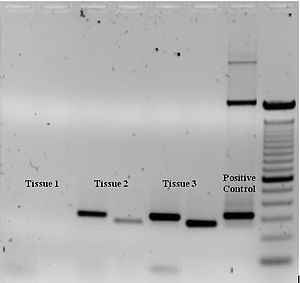
Pour v??rifier si le PCR a g??n??r?? le fragment d'ADN pr??vu (parfois d??nomm?? amplimer ou amplicon), une ??lectrophor??se sur gel d'agarose est utilis?? pour la s??paration par taille des produits de PCR. La taille (s) de produits de PCR est d??termin??e par comparaison avec une ??chelle d'ADN (un marqueur de poids mol??culaire), qui contient des fragments d'ADN de taille connue, ex??cuter sur le gel ?? c??t?? des produits de PCR (voir Fig. 3).
Stades PCR
Le proc??d?? de PCR peut ??tre divis?? en trois ??tapes:
Amplification exponentielle: A chaque cycle, la quantit?? de produit est doubl??e (en supposant 100% d'efficacit?? de la r??action). La r??action est tr??s sp??cifique et pr??cis.
Mise ?? niveau hors de la sc??ne: la r??action ralentit aussi l'ADN polym??rase perd son activit?? et que la consommation de r??actifs tels que dNTP et des amorces les am??ne ?? devenir limitante.
Plateau: Pas plus produit se accumule en raison de l'??puisement des r??actifs et des enzymes.
Optimisation PCR
Dans la pratique, la PCR peut ??chouer pour diverses raisons, en partie en raison de sa sensibilit?? ?? la contamination de produits provoquant l'amplification d'ADN parasites. De ce fait, un certain nombre de techniques et proc??dures ont ??t?? d??velopp??es pour l'optimisation des conditions de PCR. La contamination par de l'ADN ??tranger est adress??e aux protocoles et proc??dures qui s??parent m??langes pr??-PCR ?? partir d'ADN contaminants potentiels laboratoire. Cela implique g??n??ralement la s??paration spatiale des zones PCR-configuration des zones pour l'analyse ou la purification des produits de PCR, et nettoyer ?? fond la surface de travail entre les configurations de r??action. Primer techniques de conception sont importantes pour am??liorer le rendement en produit de PCR et en ??vitant la formation de produits parasites, et l'utilisation de composants tampon de remplacement ou des enzymes polymerases peuvent contribuer ?? l'amplification des r??gions longues ou autrement probl??matiques de l'ADN.
Application de la PCR
Isolement de l'ADN g??nomique
PCR permet l'isolement de fragments d'ADN de l'ADN g??nomique par amplification s??lective d'une r??gion sp??cifique de l'ADN. Cette utilisation de la PCR augmente de nombreuses m??thodes, telles que la g??n??ration sondes d'hybridation pour Sud ou l'hybridation du Nord et clonage de l'ADN, ce qui n??cessite de plus grandes quantit??s d'ADN, ce qui repr??sente une r??gion sp??cifique de l'ADN. PCR fournit ces techniques avec des quantit??s ??lev??es d'ADN pur, permettant l'analyse d'??chantillons d'ADN, m??me de tr??s petites quantit??s de mati??re de d??part.
D'autres applications de PCR comprennent Le s??quen??age d'ADN pour d??terminer des s??quences de PCR amplifi?? inconnus dans lequel l'une des amorces d'amplification peuvent ??tre utilis??es dans le s??quen??age de Sanger, l'isolement d'une s??quence d'ADN ?? acc??l??rer technologies d'ADN recombinant comportant l'insertion d'une s??quence d'ADN dans un plasmide ou le mat??riel g??n??tique d'un autre organisme. Les colonies bact??riennes ( E. coli) peut ??tre projet?? rapidement par PCR pour l'ADN correcte constructions de vecteur. PCR peut ??galement ??tre utilis??e pour empreintes g??n??tiques; une technique de m??decine l??gale utilis?? pour identifier une personne ou organisme en comparant des ADN par diff??rentes m??thodes exp??rimentales bas??es sur la PCR.
M??thodes ??empreintes digitales?? Certains PCR ont haute pouvoir discriminant et peuvent ??tre utilis??s pour identifier les relations g??n??tiques entre les individus, comme parent-enfant ou entre fr??res et s??urs, et sont utilis??s dans les tests de paternit?? (Fig. 4). Cette technique peut ??galement ??tre utilis??e pour d??terminer les relations ??volutives entre les organismes.

L'amplification et la quantification de l'ADN
Parce PCR amplifie les r??gions de l'ADN qu'il cible, la PCR peut ??tre utilis??e pour analyser de tr??s petites quantit??s d'??chantillons. Ce est souvent critique pour analyse m??dico-l??gale, alors que seulement une trace d'ADN est disponible ?? titre de preuve. PCR peut ??galement ??tre utilis?? dans l'analyse de l'ADN ancien qui est des milliers d'ann??es. Ces techniques bas??es sur la PCR ont ??t?? utilis??es avec succ??s sur des animaux, tels que quarante mille ans de mammouth , et aussi sur l'ADN humain, dans des applications allant de l'analyse des ??gyptiens momies ?? l'identification d'une Russie Tsar.
M??thodes de PCR quantitatives permettent l'estimation de la quantit?? d'une s??quence donn??e pr??sente dans un ??chantillon - une technique souvent appliqu??e pour d??terminer quantitativement les niveaux de l'expression du g??ne. PCR en temps r??el est mis en place un outil pour la quantification de l'ADN qui mesure l'accumulation de produit d'ADN apr??s chaque cycle d'amplification PCR.
- Voir ??galement L'utilisation de l'ADN dans l'entomologie m??dico-l??gale
PCR dans le diagnostic de maladies
PCR permet un diagnostic pr??coce des maladies malignes comme la leuc??mie et les lymphomes, qui est actuellement le plus d??velopp?? dans la recherche sur le cancer et est d??j?? utilis?? r??guli??rement. Des dosages de PCR peuvent ??tre r??alis??es directement sur des ??chantillons d'ADN g??nomique ?? d??tecter des cellules malignes sp??cifiques ?? translocation ?? une sensibilit?? qui est au moins 10 000 fois plus ??lev??e que les autres m??thodes.
PCR permet ??galement l'identification des micro-organismes non cultivables ou ?? croissance lente tels que les mycobact??ries, des bact??ries ana??robies ou des virus provenant de dosages de cultures tissulaires et des mod??les animaux. La base de PCR pour des applications de diagnostic en microbiologie est la d??tection d'agents infectieux et de la discrimination de non-pathog??ne de souches pathog??nes en vertu de g??nes sp??cifiques.
Viral DNA peut ??galement ??tre d??tect?? par PCR. Les amorces utilis??es doivent ??tre sp??cifiques pour des s??quences cibles dans l'ADN d'un virus, et la PCR peuvent ??tre utilis??es pour des analyses de diagnostic ou de s??quen??age de l'ADN du g??nome viral. La grande sensibilit?? de la PCR permet la d??tection des virus peu apr??s l'infection, et m??me avant le d??but de la maladie. Cette d??tection pr??coce peut donner aux m??decins une avance significative dans le traitement. La quantit?? de virus ("charge virale") chez un patient peut ??galement ??tre quantifi??e par des techniques d'ADN quantification bas??es sur la PCR (voir ci-dessous).
Des variantes de la technique PCR de base
- PCR all??le-sp??cifique: Cette technique de diagnostic ou de clonage est utilis?? pour identifier ou exploiter polymorphismes d'un seul nucl??otide (SNP) (diff??rences de base unique dans l'ADN). Il n??cessite une connaissance pr??alable de la s??quence d'ADN, y compris les diff??rences entre all??les, et utilise des amorces dont extr??mit??s 3 'englober la SNP. L'amplification par PCR dans des conditions stringentes est beaucoup moins efficace en pr??sence d'un m??sappariement entre matrice et l'amorce, de sorte amplification r??ussie d'un signal d'amorces sp??cifique SNP-SNP de la pr??sence d'une s??quence sp??cifique. Voir G??notypage SNP pour plus d'informations.
- Assembl??e PCR ou Polymerase Assembl??e Cyclisme (PCA): Assemblage PCR est la synth??se artificielle de longues s??quences d'ADN en effectuant une PCR sur un bassin de longs oligonucleotides avec de courts segments qui se chevauchent. Les oligonucl??otides alternent entre directions sens et antisens, et les segments se chevauchent d??terminent l'ordre des fragments PCR produisant ainsi s??lectivement le produit final d'ADN longtemps.
- La PCR asym??trique: PCR asym??trique est utilis??e pour amplifier pr??f??rentiellement un brin de l'original ADN plus que l'autre. Il trouve une utilisation dans certains types de s??quen??age et l'hybridation de sondage o?? avoir un seul des deux brins compl??mentaires est n??cessaire. PCR est effectu??e comme d'habitude, mais avec un grand exc??s des amorces pour le volet choisi. En raison de la lente ( arithm??tique ) amplification ult??rieure dans la r??action apr??s l'amorce limitante est ??puis??e, les cycles suppl??mentaires de PCR sont n??cessaires. Une modification r??cente sur ce processus, connu sous le nom L inear- Un fter- T il- E xponential-PCR (PCR LATE), utilise une amorce limitante avec une temp??rature de fusion sup??rieure ( Temp??rature de fusion | Tm) de l'amorce en exc??s pour maintenir l'efficacit?? de la r??action lorsque la concentration en amorce limitante diminue mi-r??action.
- H??licase d??pendante de l'amplification: Cette technique est similaire ?? une PCR classique, mais utilise plut??t une temp??rature constante de v??lo ?? travers des cycles de d??naturation et d'annelage / extension. ADN h??licase, une enzyme qui se d??roule ?? l'ADN, est utilis??e ?? la place de la d??naturation thermique.
- Hot-Start PCR: Ce est une technique qui r??duit l'amplification non sp??cifique au cours de la configuration initiale ??tapes de la PCR. La technique peut ??tre r??alis??e manuellement en chauffant les composants de la r??action ?? la temp??rature de fusion (par exemple, 95 ?? C) avant d'ajouter la polymerase. Syst??mes enzymatiques sp??cialis??s ont ??t?? mis au point qui inhibent l'activit?? de la polym??rase ?? temp??rature ambiante, soit par la liaison d'un anticorps ou par la pr??sence d'inhibiteurs li??s par covalence qui ne se dissocient apr??s une ??tape d'activation ?? haute temp??rature. D??marrage ?? chaud / froid finition PCR est r??alis??e avec de nouvelles polym??rases hybrides qui sont inactifs ?? la temp??rature ambiante et sont instantan??ment activ??es ?? la temp??rature d'??longation.
- Intersequence sp??cifique (ISSR) PCR: une m??thode PCR pour l'ADN des empreintes digitales qui amplifie les r??gions entre certaines r??p??titions de s??quences simples pour produire une empreinte digitale unique de longueur des fragments amplifi??s.
- PCR inverse: une m??thode utilis??e pour permettre PCR lorsque seulement une s??quence interne est connue. Ceci est particuli??rement utile pour identifier des s??quences flanquantes aux divers inserts g??nomiques. Il se agit d'une s??rie de digestions d'ADN et auto-ligature, r??sultant en des s??quences connues aux deux extr??mit??s de la s??quence inconnue.
- La ligature de PCR m??di??e par: Cette m??thode utilise de petits lieurs d'ADN li?? ?? l'ADN d'int??r??t et de multiples amorces recuit ?? des lieurs d'ADN; il a ??t?? utilis?? pour le s??quen??age d'ADN, le g??nome marche, et ADN empreinte.
- Sp??cifique de la m??thylation PCR (MSP): La m??thode MSP a ??t?? d??velopp?? par Stephen Baylin et Jim Herman ?? l'??cole de m??decine Johns Hopkins, et est utilis?? pour d??tecter la m??thylation des ??lots CpG dans l'ADN g??nomique. ADN est d'abord trait?? avec du bisulfite de sodium, qui convertit des bases cytosine non m??thyl??es en uracile, qui est reconnu par des amorces de PCR comme thymine. Deux PCR sont ensuite effectu??s sur l'ADN modifi??, en utilisant des ensembles d'amorces identiques sauf ?? des ??lots CpG dans les s??quences d'amorces. A ces points, une s??rie d'amorces reconna??t ADN avec cytosines pour amplifier l'ADN m??thyl??, et un ensemble reconna??t l'ADN avec de l'uracile ou la thymine pour amplifier l'ADN non m??thyl??. MSP utilisant qPCR peut ??galement ??tre effectu??e pour obtenir des informations quantitatives plut??t que qualitatives sur la m??thylation.
- Miniprimer PCR: PCR Miniprimer utilise une nouvelle polymerase thermostable (S-ROC) qui peut se ??tendre ?? partir de courtes amorces ("smalligos??) aussi court que 9 ou 10 nucleotides, au lieu des 20 nucleotides environ requises par Taq. Cette m??thode permet de PCR ciblant les r??gions de liaison d'amorce plus petit, et est particuli??rement utile pour amplifier inconnue, mais conserv??e, des s??quences d'ADN, tels que 16S ou 18S (eucaryotes) g??ne de l'ARNr. ARNr 16S miniprimer PCR a ??t?? utilis??e pour caract??riser une communaut?? de tapis microbiens croissante dans un environnement extr??me, un ??tang hypersalin ?? Puerto Rico. Dans cette ??tude, profond??ment s??quences divergentes ont ??t?? d??couverts ?? haute fr??quence et des repr??sentants que d?? fi ni deux nouveaux taxons au niveau de la division, ce qui sugg??re que la PCR miniprimer peut r??v??ler de nouvelles dimensions de la diversit?? microbienne. En ??largissant "l'espace de s??quence?? qui peut ??tre interrog?? par des amorces de PCR, cette technique peut permettre de nouvelles strat??gies de PCR qui ne sont pas possibles dans les limites de conception d'amorce impos??es par Taq et d'autres enzymes couramment utilis??es.
- Multiplex ligature d??pendant Probe Amplification (MLPA): permet de multiples cibles ?? amplifier avec seulement une seule paire d'amorces, ??vitant ainsi les limites de r??solution de PCR multiplex (voir ci-dessous).
- Multiplex PCR: L'utilisation de multiples jeux d'amorces uniques au sein d'un m??lange unique de PCR pour produire amplicons de tailles variables sp??cifiques pour diff??rentes s??quences d'ADN. En ciblant de multiples g??nes ?? la fois, des informations suppl??mentaires peuvent ??tre obtenues ?? partir d'un simple test qui n??cessiteraient autrement plusieurs fois les r??actifs et plus de temps ?? effectuer. Les temp??ratures d'hybridation pour chacun des ensembles d'amorces doivent ??tre optimis??es pour fonctionner correctement dans une seule r??action, et tailles des amplicons, ce est ?? dire, la longueur de la paire de bases, doivent ??tre suffisamment diff??rents pour former des bandes distinctes lorsque visualis?? par ??lectrophor??se sur gel.
- PCR nich??e: augmente la sp??cificit?? de l'amplification d'ADN, en r??duisant fond d?? ?? l'amplification non sp??cifique de l'ADN. Deux jeux d'amorces sont utilis??es dans deux PCR successives. Dans la premi??re r??action, une paire d'amorces est utilis??e pour g??n??rer des produits d'ADN qui, en plus de la cible vis??e, peut encore consister en des fragments d'ADN amplifi??s de mani??re non sp??cifique. Le produit (s) sont ensuite utilis??s dans une seconde PCR avec un jeu d'amorces dont les sites de liaison sont compl??tement ou partiellement diff??rente de et situ?? en 3 'de chacune des amorces utilis??es dans la premi??re r??action. PCR nich??e est souvent plus r??ussi ?? amplifier sp??cifiquement longs fragments d'ADN que la PCR conventionnelle, mais il n??cessite une connaissance plus d??taill??e des s??quences cibles.
- PCR de chevauchement-extension: est un technique de g??nie g??n??tique permettant la construction d'une s??quence d'ADN avec une alt??ration ins??r?? au-del?? de la limite de la plus grande longueur de l'amorce pratique.
- PCR quantitative (Q-PCR): est utilis?? pour mesurer la quantit?? d'un produit PCR (de pr??f??rence en temps r??el). Ce est la m??thode de choix pour mesurer quantitativement des quantit??s de d??part de l'ADN, un ADNc ou un ARN. Q-PCR est g??n??ralement utilis?? pour d??terminer si une s??quence d'ADN est pr??sente dans un ??chantillon et le nombre de ses copies dans l'??chantillon. La m??thode avec actuellement le plus haut niveau de pr??cision est la PCR quantitative en temps r??el. Il est souvent connu sous le nom de pr??ter ?? confusion RT-PCR (R eal T ime PCR) ou RQ-PCR. QRT-PCR ou RTQ-PCR sont des contractions plus appropri??s. RT-PCR se r??f??re commun??ment ?? transcription inverse PCR (voir ci-dessous), qui est souvent utilis?? en conjonction avec Q-PCR. M??thodes QRT-PCR utilisent des colorants fluorescents, tels que les SYBR Green, ou des sondes d'ADN contenant un fluorophore, tels que TaqMan, pour mesurer la quantit?? de produit amplifi?? en temps r??el.
- RT-PCR: (R??verse T ranscription PCR) est une m??thode utilis??e pour amplifier, isoler ou identifier une s??quence connue d'un cellulaire ou tissu ARN. La PCR est pr??c??d??e d'une r??action en utilisant transcriptase inverse pour convertir ARN ?? ADNc. RT-PCR est largement utilis?? dans le profilage d'expression, afin de d??terminer l'expression d'un g??ne ou d'identifier la s??quence d'un transcrit d'ARN, y compris le d??marrage de la transcription et des sites de terminaison et, si la s??quence d'ADN g??nomique d'un g??ne est connue, pour cartographier l'emplacement du exons et introns dans le g??ne. L'extr??mit?? 5 'd'un g??ne (par rapport au site d'initiation de la transcription) est g??n??ralement identifi?? par un proc??d?? de RT-PCR, nomm??e RACE-PCR, court pour l'amplification rapide des extr??mit??s d'ADNc.
- Solid Phase PCR: englobe de multiples significations, y compris Polony Amplification (o?? les colonies PCR sont d??riv??s dans une matrice de gel, par exemple), 'Bridge PCR ??(les seules amorces pr??sente sont li??s de mani??re covalente ?? la surface du support solide), en phase solide classique PCR (o?? PCR asym??trique est appliqu??e en pr??sence de solide soutien amorce de roulement avec une s??quence correspondant ?? l'une des amorces aqueuses) et Enhanced solide PCR phase (o?? phase solide conventionnelle PCR peut ??tre am??lior??e en utilisant haute Tm amorce de support solide avec application d'une '??tape' thermique ?? favoriser support solide amor??age).
- TAIL-PCR: PCR asym??trique thermique entrelac??e est utilis??e pour isoler une s??quence inconnue flanquant une s??quence connue. Dans la s??quence connue TAIL-PCR utilise une paire d'amorces imbriqu??e avec diff??rentes temp??ratures de recuit; une amorce d??g??n??r??e est utilis??e pour amplifier dans l'autre direction ?? partir de la s??quence inconnue.
- Touchdown PCR: une variante de la PCR qui vise ?? r??duire de fond non sp??cifique en abaissant progressivement la temp??rature de recuit que le cyclisme PCR progresse. La temp??rature de recuit ?? des premiers cycles est g??n??ralement de quelques degr??s (3-5c) au-dessus de la Tm des amorces utilis??es, tout en les cycles ult??rieurs, il est de quelques degr??s (3-5c) ci-dessous l'amorce T m. Les temp??ratures plus ??lev??es donnent une plus grande sp??cificit?? pour la liaison ?? l'amorce, et les temp??ratures inf??rieures permettent une amplification plus efficace des produits sp??cifiques form??s pendant les cycles initiaux.
- PAN-AC: Cette m??thode utilise pour l'amplification des conditions isothermes, et peut ??tre utilis?? dans les cellules vivantes.
- Universal marche rapide: cette m??thode permet marche du g??nome et l'empreinte g??n??tique en utilisant un plus sp??cifique ??recto-verso?? PCR que les approches conventionnelles ??unilat??rales?? (en utilisant seulement une amorce sp??cifique du g??ne et une amorce g??n??rale - qui peut conduire ?? ??bruit artefactuelle ') en vertu d'un m??canisme impliquant la formation de structure de type lariat. Rationalisation des d??riv??s de UFW sont Lane RAGE (lasso termine d??pendant PCR nich??e pour l'amplification rapide de l'ADN g??nomique), 5'RACE Lane et 3'RACE Lane.
Histoire
Un document 1971 dans la Journal of Molecular Biology par Kleppe et collaborateurs d??crit un premier proc??d?? utilisant un dosage enzymatique pour r??pliquer une matrice d'ADN avec des amorces courte in vitro. Toutefois, cette manifestation pr??coce du principe de base de PCR n'a pas re??u beaucoup d'attention, et l'invention de la r??action en cha??ne de la polym??rase en 1983 est g??n??ralement cr??dit?? ?? Kary Mullis.
Au c??ur de la m??thode PCR est l'utilisation d'un convenable ADN polym??rase capable de r??sister aux hautes temp??ratures de> 90 ?? C (> 195 ?? F) requise pour la s??paration des deux brins d'ADN dans le Double h??lice de l'ADN apr??s chaque cycle de r??plication. Les ADN polymerases utilis??es initialement pour pr??sageant exp??riences in vitro par PCR ont ??t?? incapables de r??sister ?? ces temp??ratures ??lev??es. Ainsi, les premi??res proc??dures de replication de l'ADN ont ??t?? tr??s inefficaces, de temps, et doivent de grandes quantit??s d'ADN polymerase et de manutention continue tout au long du processus.
Une d??couverte de 1976 Une polymerase Taq ADN polym??rase purifi??e ?? partir de la bact??rie thermophile, Thermus aquaticus, qui se produit naturellement dans l'eau chaude (50-80 ?? C (120-175 ?? F)) environnements ouvert la voie ?? des am??liorations spectaculaires de la m??thode PCR. La polymerase d'ADN isol?? ?? partir de T. aquaticus est stable ?? des temp??ratures ??lev??es en restant actif m??me apr??s d??naturation de l'ADN, ce qui ??vite la n??cessit?? d'ajouter de nouveaux ADN polym??rase apr??s chaque cycle. Cela a permis un processus fond?? de thermocycleur-automatis?? pour l'amplification d'ADN.
Au moment o?? il a d??velopp?? PCR en 1983, travaillait dans Mullis Emeryville, en Californie, pour Cetus Corporation, l'un des premiers biotechnologie entreprises. L??, il ??tait responsable de la synth??se de courtes cha??nes d'ADN. Mullis a ??crit qu'il a con??u de la PCR en croisi??re le long de la Pacific Coast Highway une nuit dans sa voiture. Il jouait dans son esprit avec une nouvelle fa??on d'analyser les changements (mutations) dans l'ADN quand il a r??alis?? qu'il avait plut??t invent?? un proc??d?? d'amplification d'une r??gion de l'ADN par des cycles r??p??t??s de duplication entra??n??e par l'ADN polym??rase.
En Scientific American, Mullis r??sum?? la proc??dure: "A partir d'une seule mol??cule de l'ADN du mat??riel g??n??tique, le PCR peut g??n??rer 100 milliards de mol??cules semblables dans un apr??s-midi La r??action est facile ?? ex??cuter Il ne n??cessite pas plus d'un tube ?? essai, quelques-uns.. r??actifs simples, et une source de chaleur. " Il a re??u le prix Nobel de chimie en 1993 pour son invention, sept ans apr??s que lui et ses coll??gues de Cetus premi??re mise sa proposition ?? la pratique. Cependant, certaines controverses sont rest??s sur les contributions intellectuelles et pratiques d'autres scientifiques aux travaux de Mullis, et se il avait ??t?? le seul inventeur du principe PCR. (Voir l'article principal: Kary Mullis)
Guerre des brevets
La technique PCR a ??t?? brevet?? par Cetus Corporation, o?? Mullis a travaill?? quand il a invent?? la technique en 1983. L'enzyme Taq polym??rase a ??galement ??t?? couverts par des brevets. Il ya eu plusieurs proc??s tr??s m??diatis??s li??s ?? la technique, y compris une poursuite intent??e par infructueuse DuPont. La compagnie pharmaceutique Hoffmann-La Roche a achet?? les droits sur les brevets en 1992 et d??tient actuellement ceux qui sont encore prot??g??s.
Une bataille de brevet li??es sur l'enzyme Taq polym??rase est toujours en cours dans plusieurs pays ?? travers le monde entre Roche et Promega. Les arguments juridiques ont ??tendu au-del?? de la dur??e de vie des brevets de la polym??rase Taq PCR et originaux, qui a expir?? le 28 mars, 2005

