Centromère
|
|
Cet article est une ébauche concernant la biologie. Vous pouvez partager vos connaissances en l’améliorant (comment ?) selon les recommandations des projets correspondants.
|
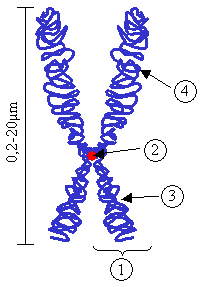
(1) Chromatide. Chacune formée d'un brin d'ADN parental et d'un brin d'ADN néoformé obtenu après réplication durant la phase S ; (2) Centromère. Le point de contact des deux chromatides, et le point de séparation lors de la mitose ; (3) Bras court ; (4) Bras long
Le centromère est la partie située au milieu des 2 chromatides.
Typologie
Il existe deux types de centromères.
Les centromères des chromosomes monocentriques sont des centromères « régionaux », occupant une région précise au sein d'un chromosome. Ils sont généralement visibles sous la forme d'une constriction sur le chromosome métaphasique. La taille de cette région varie entre les espèces, de 125 pb chez S. cerevisiae jusqu'à plusieurs mégabases pour les centromères humains[1]. On ne compte normalement qu’un centromère régional par chromosome (d’où le terme « monocentrique »), mais des réarrangements chromosomiques peuvent conduire à deux centromères sur un même chromosome (on parle alors de chromosome dicentrique)[2].
Les centromères des chromosomes holocentriques s’étendent sur toute la longueur des chromosomes. Peu fréquents chez les espèces couramment étudiées (à l’exception de C. elegans), jamais observés chez les vertébrés, les chromosomes holocentriques sont néanmoins répandus chez les protozoaires, plusieurs phylums d’invertébrés et quelques plantes[3].
La composition en ADN
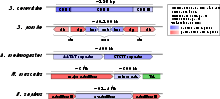
L’ADN centromérique est très variable d’une espèce à l’autre. Le centromère le plus simple, celui de Saccharomyces cerevisiae, s’étend sur environ 125 pb et est composé de trois éléments fonctionnels CDE I, II et III (Centromere DNA Element), les éléments I et III étant conservés sur les seize chromosomes alors que l’élément II a une séquence variable, unique sur chaque chromosome[1].
Chez S. pombe, les centromères ont une taille allant de ~35 kb sur le chromosome I à 110 kb sur le chromosome III[4]. Chaque centromère est composé d’un domaine central flanqué de deux domaines externes. Le domaine central est lui-même composé d’un élément central cnt de 4 à 7 kb constitué d’ADN non-répété, entouré de deux éléments d’ADN répété inversés, les éléments imr (innermost repeats). La séquence de tous les éléments du domaine central, cnt et imr, est unique sur chaque chromosome, seule l’organisation du domaine central est conservé d’un chromosome à l’autre. Les domaines externes, otr pour outer repeats, sont essentiellement composés d’un nombre variable de copies de deux éléments d’ADN répété, dg et dh.
Chez les métazoaires, l’ADN centromérique est constitué essentiellement de différentes formes d’ADN répété pouvant s’étendre sur plusieurs mégabases. Cette composition constitue un obstacle à leur analyse détaillée, le séquençage et l’assemblage de longues régions d’ADN répété étant difficile. Chez la drosophile par exemple, un seul centromère, celui du minichromosome Dp1187, a été étudié à l’échelle de la séquence nucléotidique[5]. Il s’étend sur 420 kb divisés en deux régions composées de deux types d’ADN satellite distincts entrecoupés d’éléments transposables.

Les flèches correspondent aux monomères. Les couleurs indiquent les écarts d'identité (polymorphismes) de séquences. Les HOR sont très semblables, mais les monomères au sein des HOR sont très différents.
Chez l’homme, l’ADN répété centromérique est principalement de l’ADN α-satellite (aussi appelé alphoid DNA en anglais), dont le motif unitaire (monomère) fait 171 pb[6]. Ces monomères sont organisés en motifs répétés d’ordre supérieur (higher order repeat ou HOR), alignés de manière ininterrompue dans la même orientation sur des distances allant de 100 kb à 4 Mb. Au sein d’un même chromosome, les HOR sont très homogènes, avec 99.8 % d’identité de séquence, alors que les monomères au sein d’un HOR ne partagent qu’entre 60 et 80 % d’identité. L’ADN α-satellite contient une séquence particulière, la boîte CENP-B (CENP-B Box), de 17 pb, capable de lier la protéine CENP-B et impliquée dans la formation de la chromatine centromérique et l’assemblage du kinétochore.
L’absence de conservation des ADN centromériques a amené la suggestion que la fonction du centromère et sa position sur le chromosome ne sont pas déterminées par la séquence nucléotidique[1]. À l’exception de S. cerevisiae, où les éléments CDE sus-mentionnés suffisent à former un centromère fonctionnel, l’ADN centromérique chez les autres eucaryotes n’apparaît ni nécessaire ni suffisant pour supporter la fonction du centromère : non nécessaire, car un centromère fonctionnel peut se former sur des régions n’offrant aucune similitude avec des séquences centromériques (néocentromères) ; ni suffisant, car la seule présence de séquences de type centromérique ne conduit pas systématiquement à un centromère fonctionnel. Il est aujourd’hui admis que l’identité et la fonction du centromère sont définies épigénétiquement et dépendent d’une structure chromatinienne spécialisée plutôt que de la séquence d’ADN sous-jacente. Contrairement à la séquence d’ADN, la chromatine associée aux centroméres présente des caractéristiques relativement bien conservées à travers les eucaryotes.
La chromatine centromérique
La chromatine au niveau des centromères possède des caractéristiques particulières qui la distinguent à la fois de l'euchromatine et de l'hétérochromatine, si bien qu’on ne peut la qualifier que de centromérique (parfois centrochromatine ou, surtout en anglais, chromatine CEN).
La caractéristique sans doute la plus importante, conservée à travers tous les eucaryotes étudiés, est la présence de la protéine CenH3 (centromeric H3), un variant de l'histone H3 spécifiquement et exclusivement localisé dans la chromatine centromérique[6]. Cette histone (appelée aussi Cse4p chez S. cerevisiae, Cnp1p chez S. pombe, CENP-A chez la plupart des mammifères) permet la formation d’un nucléosome variant dont la structure exacte n’est pas encore clairement établie mais est, dans tous les cas, suffisamment différente de celle d’un nucléosome conventionnel pour conférer à la chromatine centromérique des propriétés distinctes[7].
D’autres caractéristiques de la chromatine centromérique sont l’hypo-acétylation des histones (une caractéristique fréquemment associée à l’hétérochromatine) et la diméthylation de l'histone H3 sur la lysine-4 (H3K4me2, une marque fréquemment associée aux régions promotrices des gènes)[6].
La chromatine centromérique est généralement flanquée de part et d’autre par de l’hétérochromatine, qualifiée de péricentromérique ou péricentrique. Elle porte les marques typiques de l’hétérochromatine, notamment la di - et tri-méthylation de l’histone H3 sur la lysine-9 (H3K9me2/3) et la présence de la protéine HP1[6]. Elle est également enrichie en variant H2A.Z, un variant de l'histone H2A[8].
La fondation du kinétochore
La fonction essentielle du centromère est de servir de plate-forme d’assemblage pour le kinétochore lors de la mitose. La protéine CenH3 de la chromatine centromérique est l’un des facteurs clefs de cette fonction. Son absence empêche toute formation du kinétochore, et sa surexpression et sa localisation ectopique entraînent une mauvaise localisation d’une partie au moins des protéines du kinétochore[7].
CenH3 permet l’assemblage du kinétochore par l’intermédiaire des protéines du complexe CCAN (constitutive centromere-associated network, « réseau [de protéines] constitutivement associé au centromère »)[9], qui reconnaissent la structure particulière des nucléosomes contenant CenH3. Le CCAN représente l’interface entre la chromatine centromérique et le kinétochore interne et permet le recrutement des différents complexes composant le kinétochore.
Chez l'homme, le CCAN peut être divisé en deux sous-complexes : le complexe CENP-ANAC (CENP-A nucleosome associated complex, « complexe associé au nucléosome CENP-A »), qui comme son nom l’indique est directement en contact avec le nucléosome CENP-A ; et le complexe CENP-ACAD (CENP-A distal complex), qui interagit non pas directement avec CENP-A mais avec les protéines du complexe CENP-ANAC[7]. Chez S. cerevisiae et S. pombe, les homologues fonctionnels du CCAN humain sont les complexes Ctf19 et Sim4, respectivement[10]. Les homologues du CCAN chez les autres eucaryotes modèles n’ont pas encore été identifiés.
Voir aussi
Bibliographie
- (en) Ben E. Black et Emily A. Bassett, « The histone variant CENP-A and centromere specification », Curr. Opin. Cell Biol., vol. 20, no 1, , p. 91–100 (PMID 18226513, DOI 10.1016/j.ceb.2007.11.007)
- (en) Iain Cheeseman et Arshad Desai, « Molecular architecture of the kinetochore-microtubule interface », Nat. Rev. Mol. Cell Biol., vol. 9, no 1, , p. 33–46 (PMID 18097444, DOI 10.1038/nrm2310)
- (en) Don Cleveland, Yinghui Mao et Kevin Sullivan, « Centromeres and kinetochores: from epigenetics to mitotic checkpoint signaling », Cell, vol. 112, no 4, , p. 407–421 (PMID 12600307, lire en ligne)
- (en) Ian K. Greaves, Danny Rangasamy, Patricia Ridgway et David J. Tremethick, « H2A.Z contributes to the unique 3D structure of the centromere », Proc. Natl. Acad. Sci. USA, vol. 104, no 2, , p. 525–530 (PMID 17194760, DOI 10.1073/pnas.0607870104, lire en ligne)
- (en) M. Guerra, G. Cabral, M. Cuacos, M. González-García, M. González-Sánchez, J. Vega et M. Puertas, « Neocentrics and holokinetics (holocentrics): chromosomes out of the centromeric rules », Cytogenet. Genome Res., vol. 129, no 1-3, , p. 82–96 (PMID 20551611, DOI 10.1159/000314289)
- (en) Alison L. Pidoux et Robin C. Allshire, « Kinetochore and heterochromatin domains of the fission yeast centromere », Chromosome Res., vol. 12, no 6, , p. 521–534 (PMID 15289660, DOI 10.1023/B:CHRO.0000036586.81775.8b)
- (en) Stefano Santaguida et Andrea Musacchio, « The life and miracles of kinetochores », EMBO J., vol. 28, no 17, , p. 2511–2531 (PMID 19629042, DOI 10.1038/emboj.2009.173)
- (en) Mary G. Schueler et Beth A. Sullivan, « Structural and functional dynamics of human centromeric chromatin », Annu. Rev. Genomics Hum. Genet., vol. 7, , p. 301–313 (PMID 16756479, DOI 10.1146/annurev.genom.7.080505.115613)
- (en) Beth A. Sullivan et Huntington F. Willard, « Stable dicentric X chromosomes with two functional centromeres », Nat. Genet., vol. 20, no 3, , p. 227–228 (PMID 9806536, DOI 10.1038/3024)
- (en) Xiaoping Sun, Janice Wahlstrom et Gary Karpen, « Molecular structure of a functional Drosophila centromere », Cell, vol. 91, no 7, , p. 1007–1019 (PMID 9428523, lire en ligne)
Notes et références
- Portail de la biologie cellulaire et moléculaire
